obt_fab_master
Lisa
2024-03-27
Небольшой пример обработки микробных данных на примере анализа измкенчивости микробиома литторин (данные изменены)
Осторожно, встречаются неоптимизированные действия, неединообразный код и конфликты пакетов.
1 Основные действия до фильтрации
1.1 Загрузка данных
## phyloseq-class experiment-level object
## otu_table() OTU Table: [ 7920 taxa and 80 samples ]
## sample_data() Sample Data: [ 80 samples by 12 sample variables ]
## tax_table() Taxonomy Table: [ 7920 taxa by 7 taxonomic ranks ]## [1] "L1_L_fab_gut2" "L1_L_nod_BF2" "L1_L_obt_gut1" "L1_L_obt_gut2"
## [5] "L1_L_obt_gut3" "L1_L_obt_gut5" "L1_L_obt_gut6" "L1_L_obt_gut7"
## [9] "L1_L_obt_gut8" "L1_L_ser_BF1" "L1_L_ser_BF2" "L1_L_stones_BF1"
## [13] "L1_L_ves_BF1" "L1_L_ves_BF2" "L1_L_ves_BF3" "L1_U_obt_gut1"
## [17] "L1_U_obt_gut2" "L1_U_obt_gut3" "L1_U_obt_gut7" "L11_L_fab_gut2"
## [21] "L11_L_fab_gut3" "L11_L_fab_gut4" "L11_L_fab_gut5" "L11_L_fab_gut6"
## [25] "L11_L_fab_gut7" "L11_L_fab_gut8" "L11_L_fab_tent1" "L11_L_fab_tent2"
## [29] "L11_L_fill_BF1" "L11_L_fill_BF2" "L11_L_fill_BF3" "L11_L_nod_BF2"
## [33] "L11_L_nod_BF3" "L11_L_obt_gut3" "L11_L_obt_gut4" "L11_L_obt_gut5"
## [37] "L11_L_obt_gut6" "L11_L_obt_gut7" "L11_L_obt_gut8" "L11_L_obt_tent1"
## [41] "L11_L_obt_tent2" "L11_L_stones_BF1" "L11_L_stones_BF2" "L11_L_stones_BF3"
## [45] "L11_U_fill_BF1" "L11_U_nod_BF2" "L11_U_obt_gut1" "L11_U_obt_gut2"
## [49] "L11_U_obt_gut3" "L11_U_obt_gut4" "L11_U_obt_gut5" "L11_U_obt_gut8"
## [53] "L11_U_obt_tent2" "L11_U_stones_BF1" "L11_U_stones_BF3" "L5_L_fab_gut1"
## [57] "L5_L_fab_gut3" "L5_L_fab_gut5" "L5_L_fab_gut6" "L5_L_fab_gut7"
## [61] "L5_L_fab_gut8" "L5_L_nod_BF2" "L5_L_obt_gut1" "L5_L_obt_gut3"
## [65] "L5_L_obt_gut4" "L5_L_obt_gut7" "L5_L_obt_gut8" "L5_L_stones_BF2"
## [69] "L5_L_ves_BF3" "L5_U_fill_BF1" "L5_U_fill_BF2" "L5_U_nod_BF1"
## [73] "L5_U_nod_BF3" "L5_U_obt_gut1" "L5_U_obt_gut2" "L5_U_obt_gut4"
## [77] "L5_U_obt_gut5" "L5_U_obt_gut6" "L5_U_obt_gut8" "L5_U_obt_tent2"## [1] "Domain" "Phylum" "Class" "Order" "Family" "Genus" "Species"## [1] "site" "species" "sp_type"
## [4] "subtype" "type" "sufficient"
## [7] "level" "sea" "season"
## [10] "season_time" "season_site" "season_site_level"2 Фильтрация и нормализация образцов
2.1 Общая фильтрация
Фильтрация мягкая - удаляем ASV, встреченные 1 раз во всем наборе данных (singletones), ASV, присутствующие менее чем в 5% образцов. Порог обилия моджет быть иным, но даже если удалить фичи, встреченные менее, чем в 10%, это может помешать найти различия между образцами по редким микробам.
## L11_L_obt_gut5 L11_L_obt_gut7 L1_L_stones_BF1 L5_L_ves_BF3 L11_L_fill_BF2
## 0 0 2715 4108 4621
## L1_L_obt_gut7 L11_L_obt_gut6 L5_U_obt_gut2 L5_L_obt_gut7 L11_L_obt_gut3
## 5210 5366 5494 6242 6543
## L5_L_obt_gut8 L11_U_obt_gut1 L11_L_fab_tent2 L5_U_obt_gut5 L5_L_fab_gut6
## 7368 8125 8395 8413 84692.2 Удаление низкой глубины прочтения
первые два образца (с наиболее низкой глубиной прочтения, она НОЛЬ) далее в анализе не используются
кроме того, имеет смысл удалить лишние летние образцы (Лето 2021, неполные L.fabalis)
было принято волевое решение не удалять единственную пробу F.vesiculosus
## L5_L_ves_BF3 L11_L_fill_BF2 L11_L_obt_gut6 L5_U_obt_gut2 L5_L_obt_gut7
## 4108 4621 5366 5494 6242
## L11_L_obt_gut3 L5_L_obt_gut8 L11_U_obt_gut1 L11_L_fab_tent2 L5_U_obt_gut5
## 6543 7368 8125 8395 8413
## L5_L_fab_gut6 L5_L_obt_gut1 L11_L_nod_BF2 L5_L_obt_gut3 L11_L_obt_gut4
## 8469 8531 8536 8813 8861Значения размеров библиотек для самых больших образцов если что такие:
## L5_U_obt_gut1 L11_L_stones_BF1 L11_U_obt_gut3 L11_U_obt_gut5
## 15521 15837 17379 17694
## L11_U_obt_gut8 L11_L_fill_BF3 L11_U_obt_gut4 L5_L_nod_BF2
## 17959 18104 18279 18738
## L11_L_fill_BF1 L11_U_stones_BF1 L11_U_stones_BF3 L11_U_nod_BF2
## 18896 19070 19611 21035
## L5_U_nod_BF3 L11_L_stones_BF3 L11_L_stones_BF2
## 21893 22115 349882.3 Нормализация
Мы в своем анализе делили на медианный размер библиотеки (здесь - 11539) для всех образцов, но можно хоть логарифмировать

3 Интегральные характеристики
Давайте сначала опишем простые вещи
3.1 Таблица
альфа-ранообразие, полный датасет, объединено по типу образца
3.2 Выровненность и Шеннон общие руками
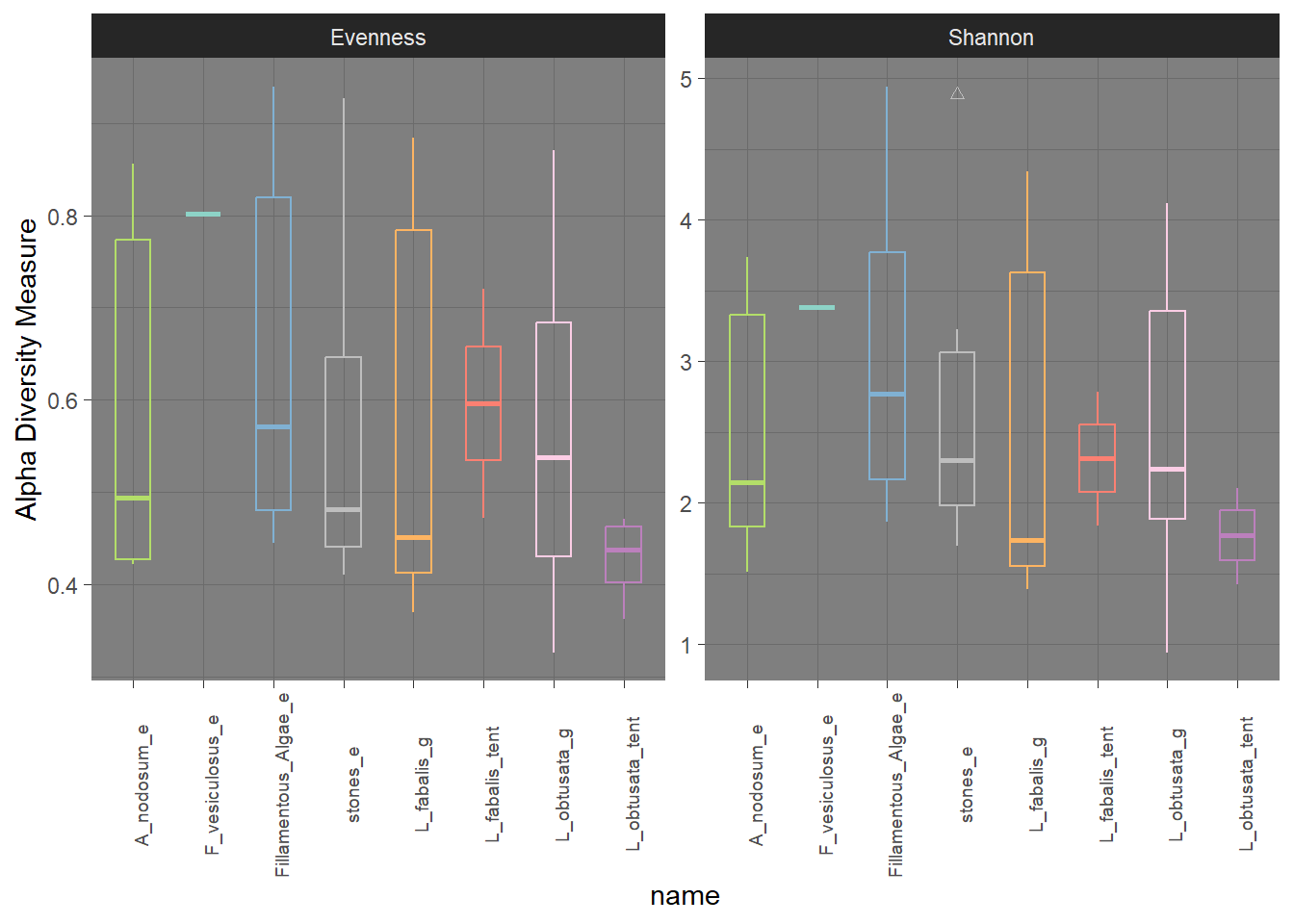
3.3 Автоматом построенный Шеннон
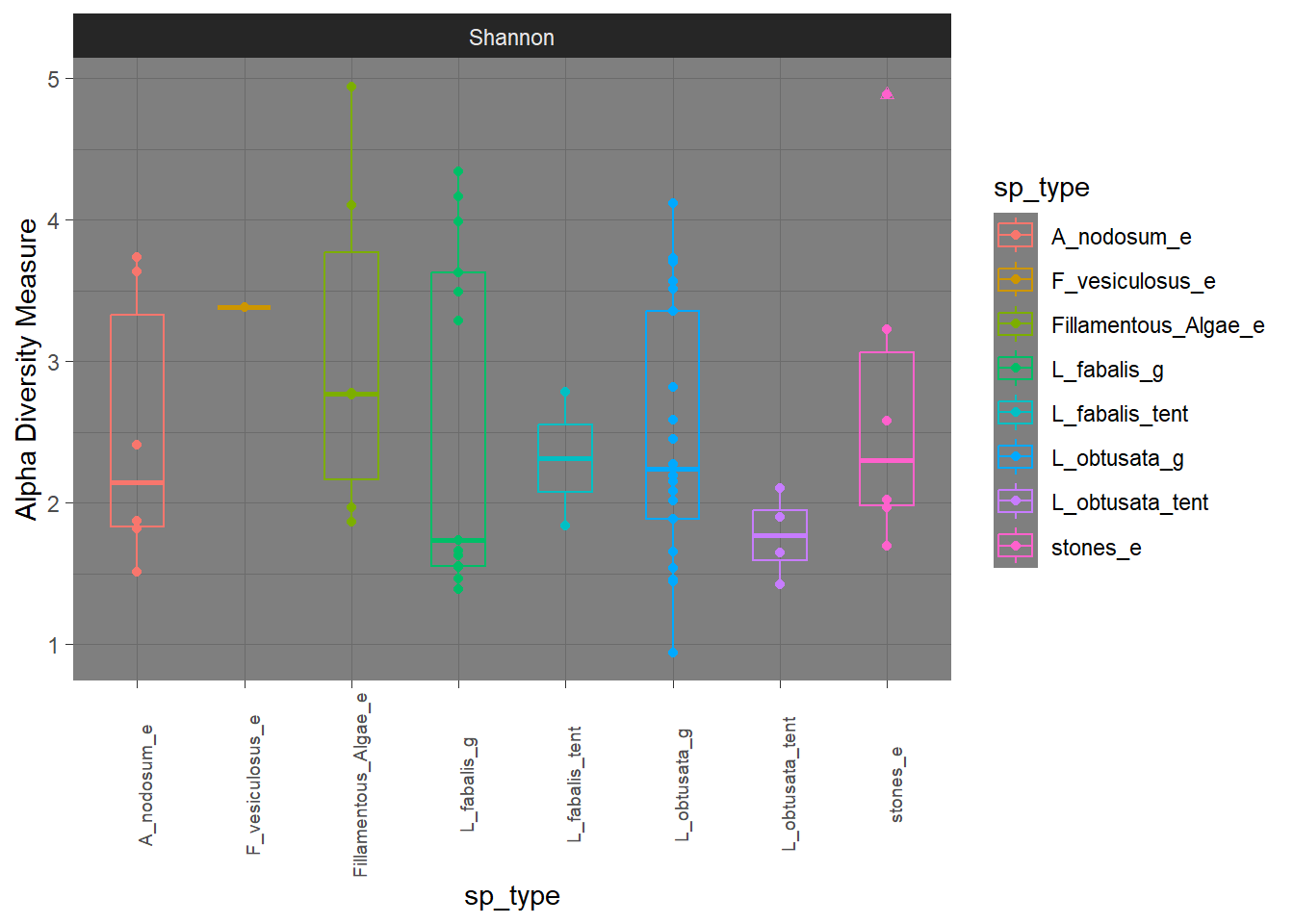
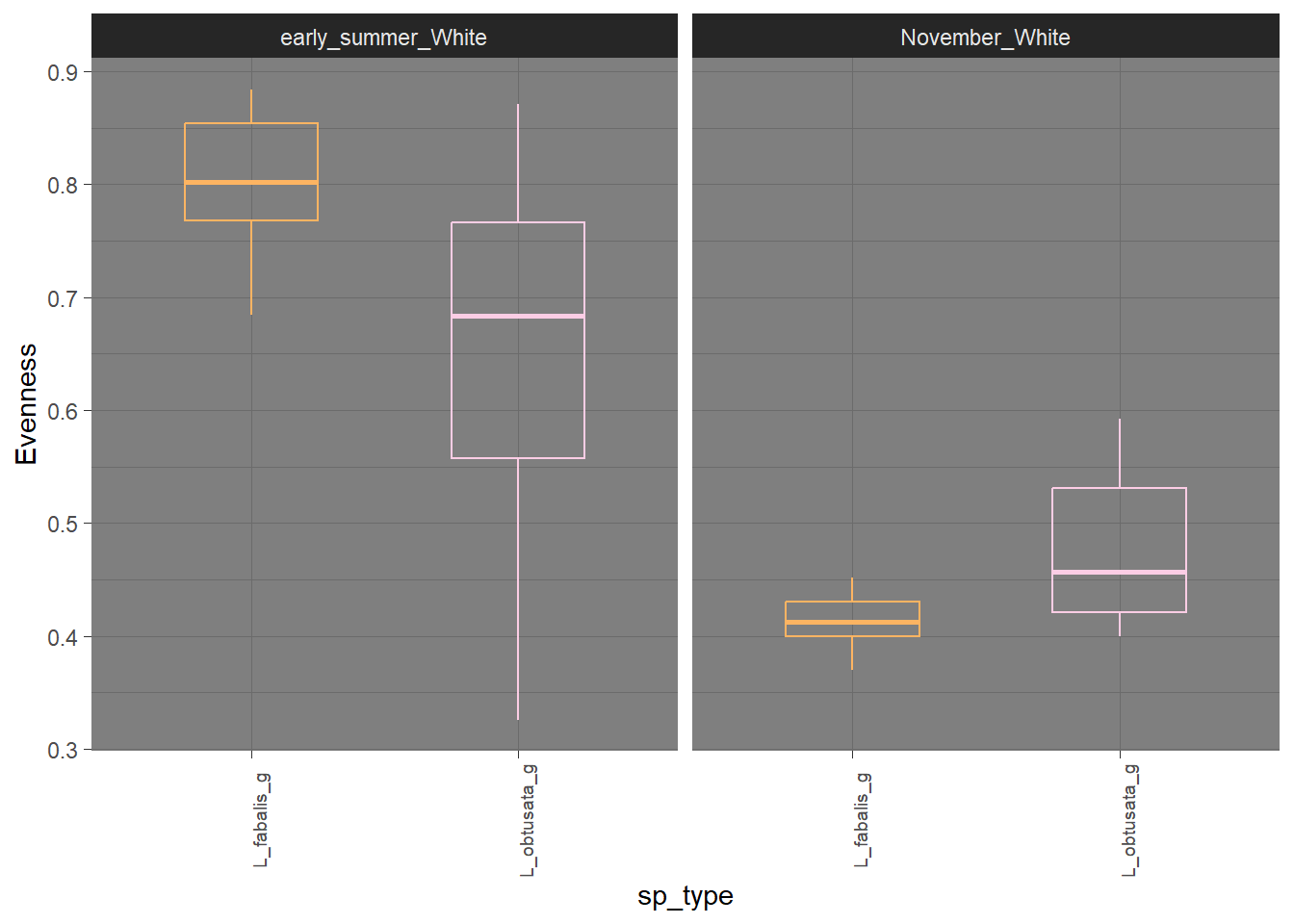
3.3.0.1 Шеннон в кишках, по сезонам на Белом
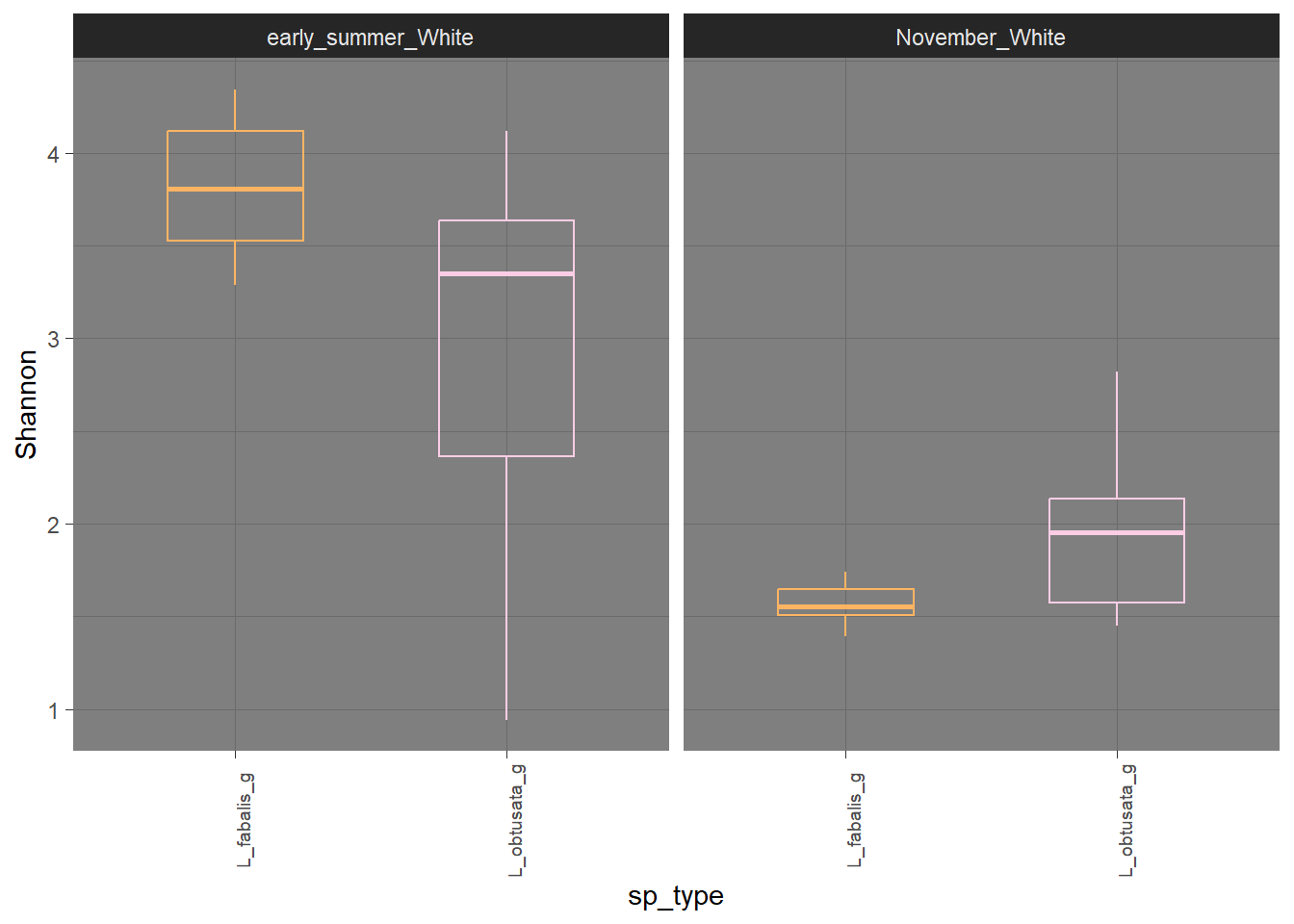
3.3.0.2 Выровненность в кишках, по сезонам на Белом
4 Ординации и PERMANOVA
Наконец-то многомерный подход
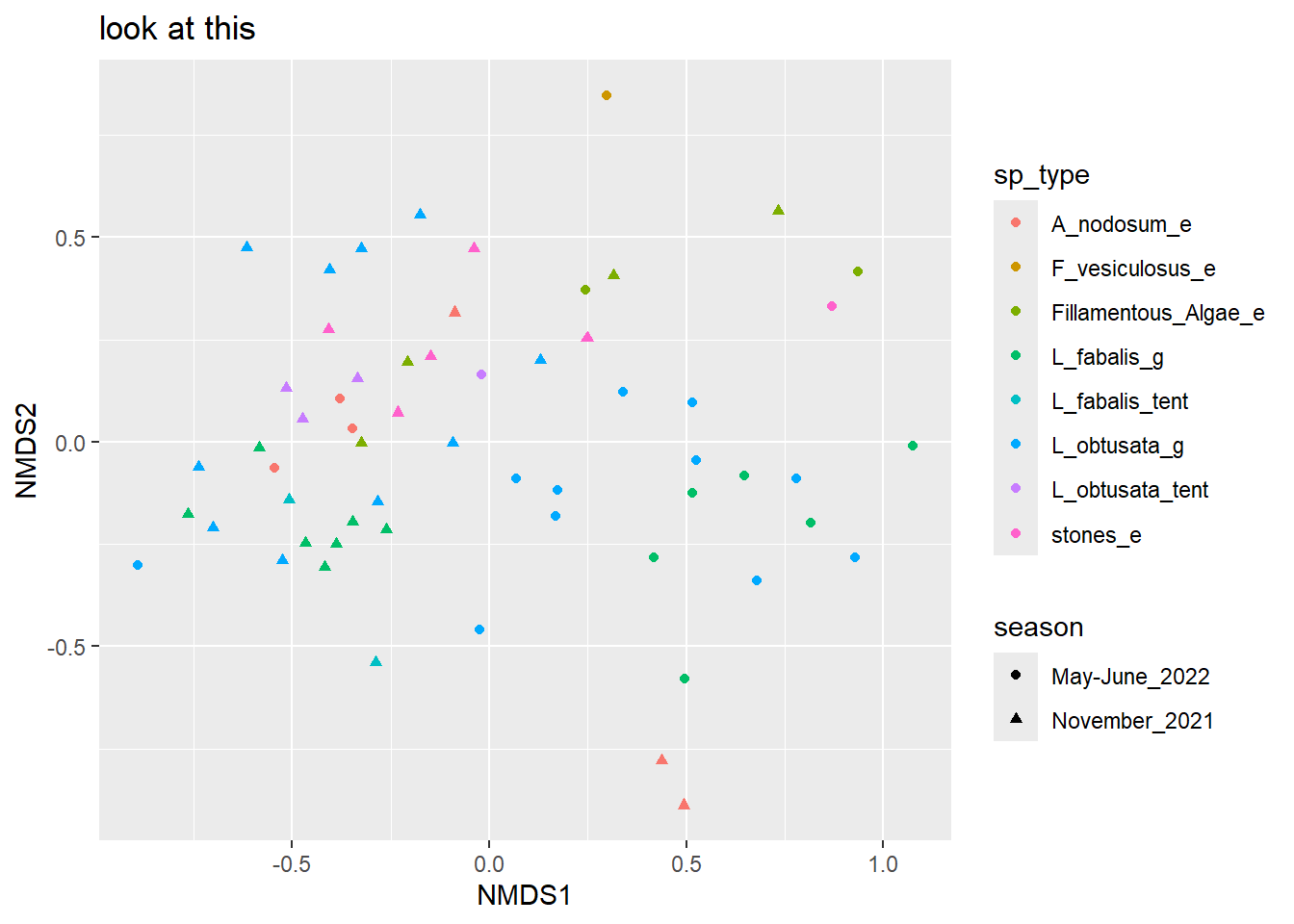
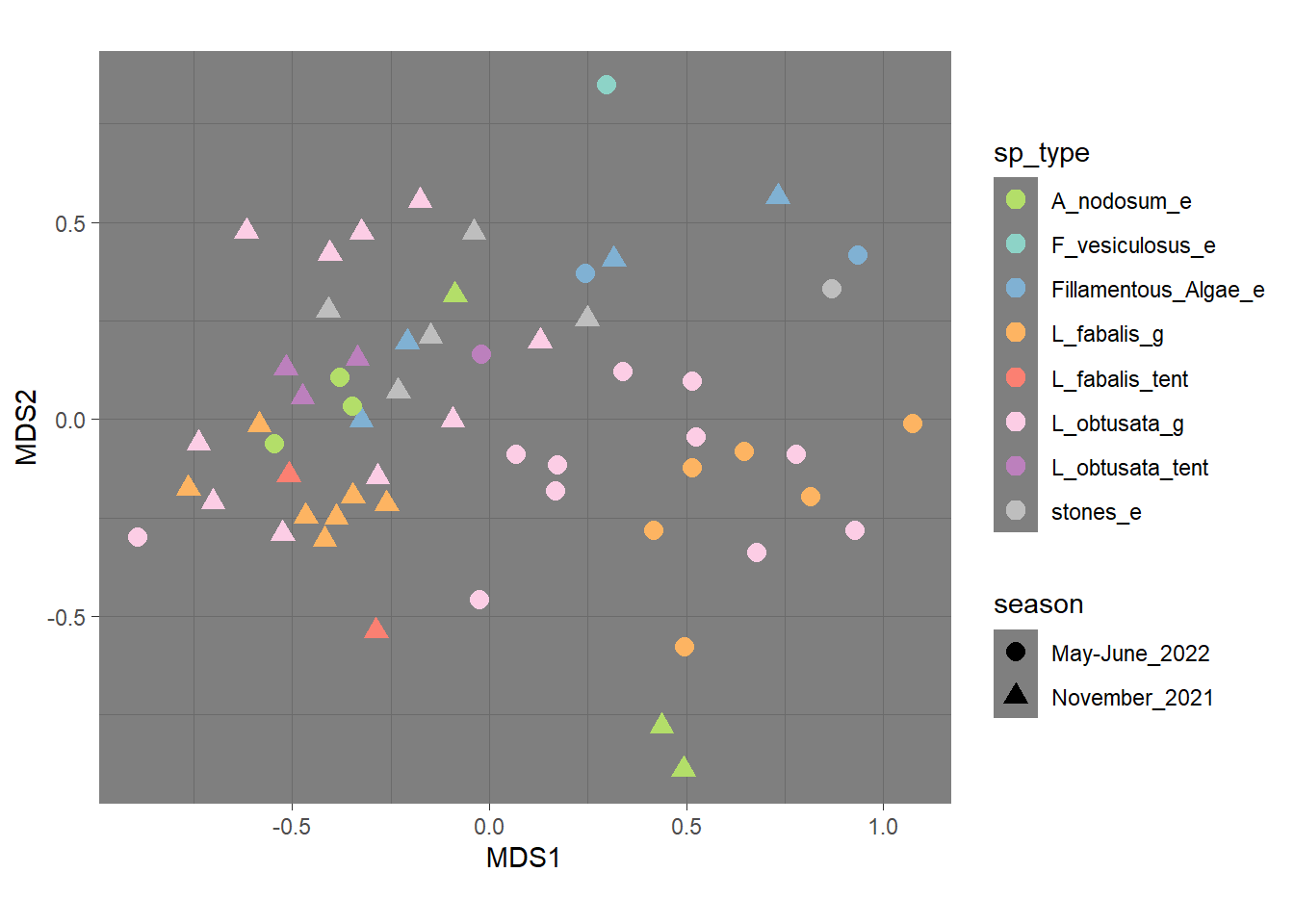
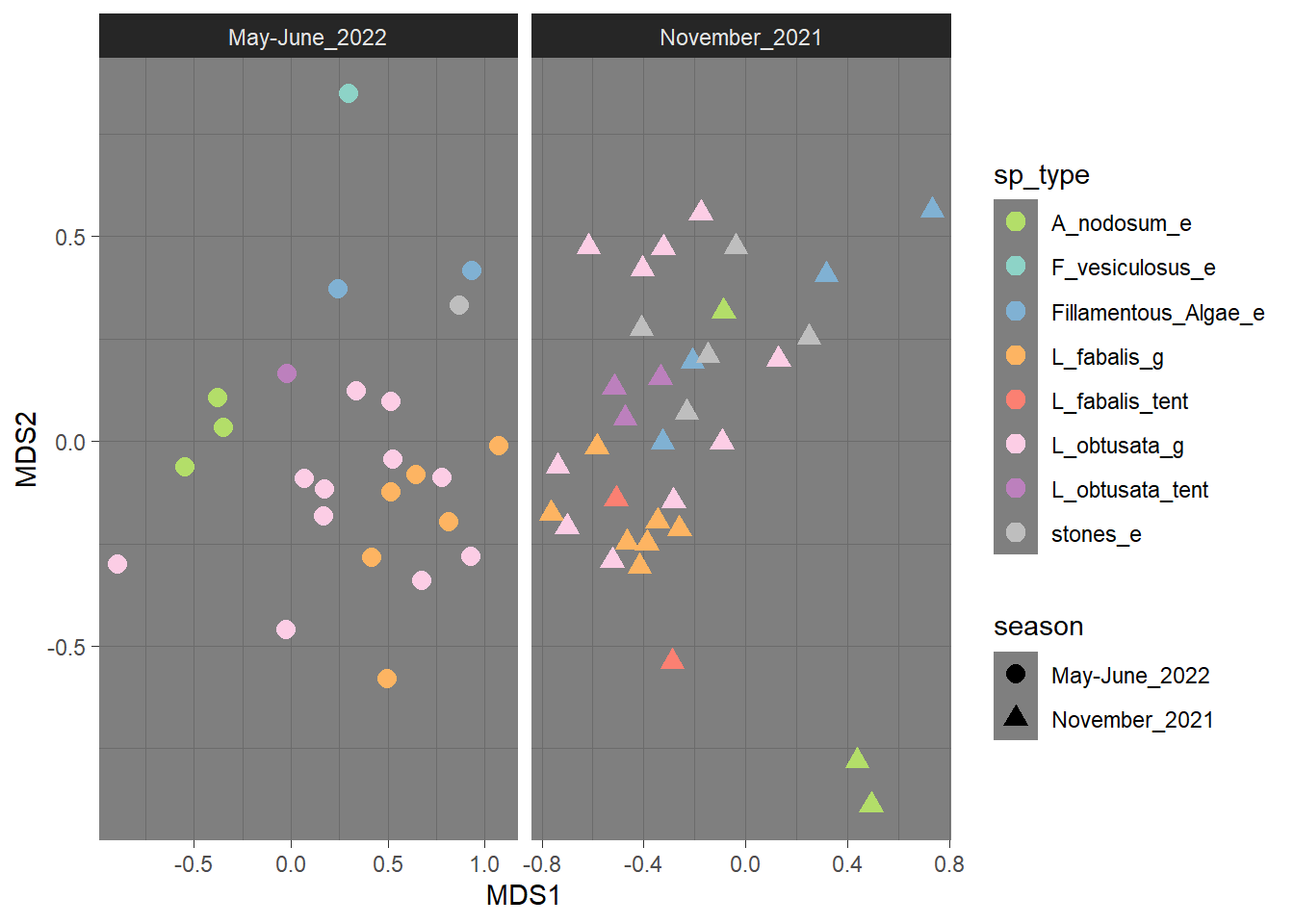
Базово я привыкла делать nMDS по матрице различий Брея-Куртиса, но с некоторыми (напр, логарифмическими нормализациями он не дружит) Можно делать и Жаккара в качестве расстояний, и хоть главные компоненты в качестве ординации Можно делать хитмап или кластеризацию, главное понять, на какие расстояния относит композиция микробиома ваши образцы друг ото друга
## Square root transformation
## Wisconsin double standardization
## Run 0 stress 0.200135
## Run 1 stress 0.2024438
## Run 2 stress 0.2051878
## Run 3 stress 0.1987549
## ... New best solution
## ... Procrustes: rmse 0.02563272 max resid 0.1652464
## Run 4 stress 0.2016256
## Run 5 stress 0.1983178
## ... New best solution
## ... Procrustes: rmse 0.01145341 max resid 0.07749063
## Run 6 stress 0.2079791
## Run 7 stress 0.2241312
## Run 8 stress 0.2095957
## Run 9 stress 0.2065654
## Run 10 stress 0.2037054
## Run 11 stress 0.1969889
## ... New best solution
## ... Procrustes: rmse 0.03064541 max resid 0.217077
## Run 12 stress 0.209418
## Run 13 stress 0.2169186
## Run 14 stress 0.2050241
## Run 15 stress 0.2060473
## Run 16 stress 0.2028711
## Run 17 stress 0.2044867
## Run 18 stress 0.2236881
## Run 19 stress 0.207731
## Run 20 stress 0.2051714
## *** Best solution was not repeated -- monoMDS stopping criteria:
## 18: stress ratio > sratmax
## 2: scale factor of the gradient < sfgrmin## [1] 0.19698894.1 Картинки
4.1.1 Все точки на одной панели автоматом
4.1.2 Все точки на одной панели руками
4.1.3 Фасетки по сезонам
4.1.4 Кишечная ординация
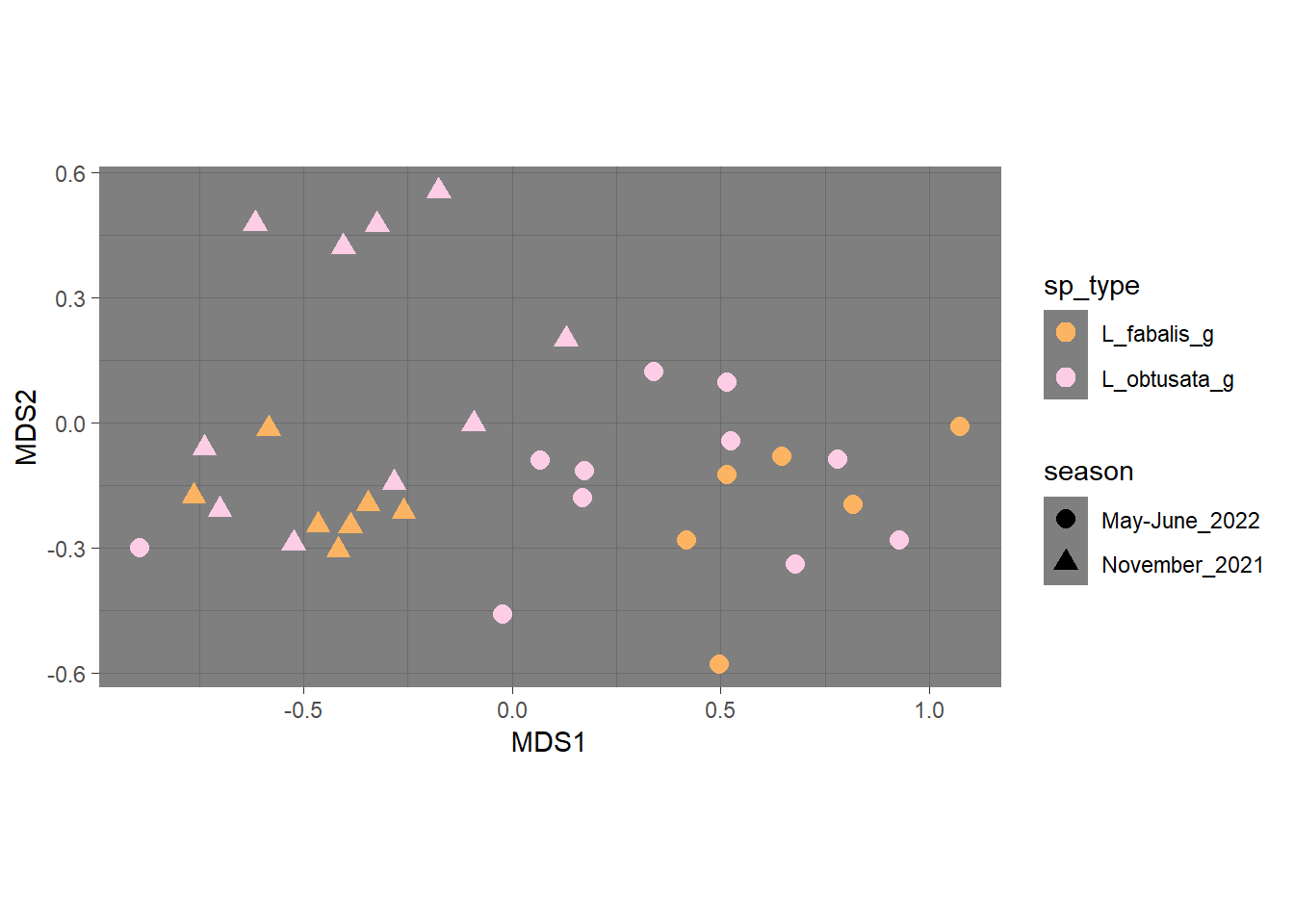
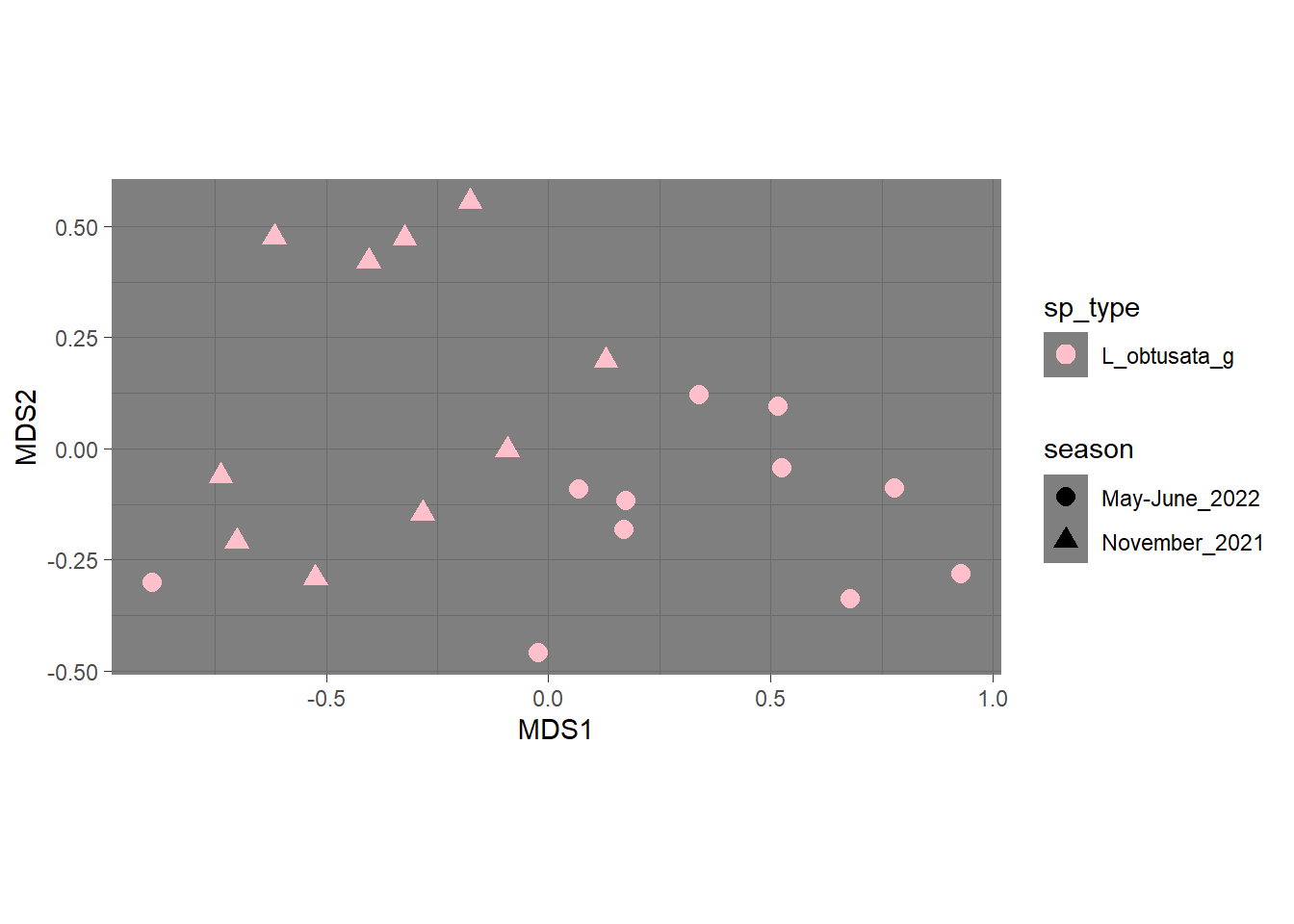
4.1.5 Для одного вида
4.2 Циферки
будем тестировать с помощью PERMANOVA, есть ли влияние вида для кишечного микробиома и есть ли влияние сезона
4.2.1 кишки, 2 сезона, 2 вида, 2 фактора
4.2.2 кишки летнего сезона, 2 вида, однофакторно
4.2.3 кишки летнего сезона, 2 вида, однофакторно, иная перманова
##
## Analysis of Variance, using Residual Randomization
## Permutation procedure: Randomization of null model residuals
## Number of permutations: 1000
## Estimation method: Ordinary Least Squares
## Sums of Squares and Cross-products: Type I
## Effect sizes (Z) based on F distributions
##
## Df SS MS Rsq F Z Pr(>F)
## species 1 0.3341 0.33409 0.09513 1.5769 1.3603 0.089 .
## Residuals 15 3.1779 0.21186 0.90487
## Total 16 3.5120
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Call: lm.rrpp(f1 = guts_white_bray_snails ~ species, data = guts_white_df)5 Барплоты обилия
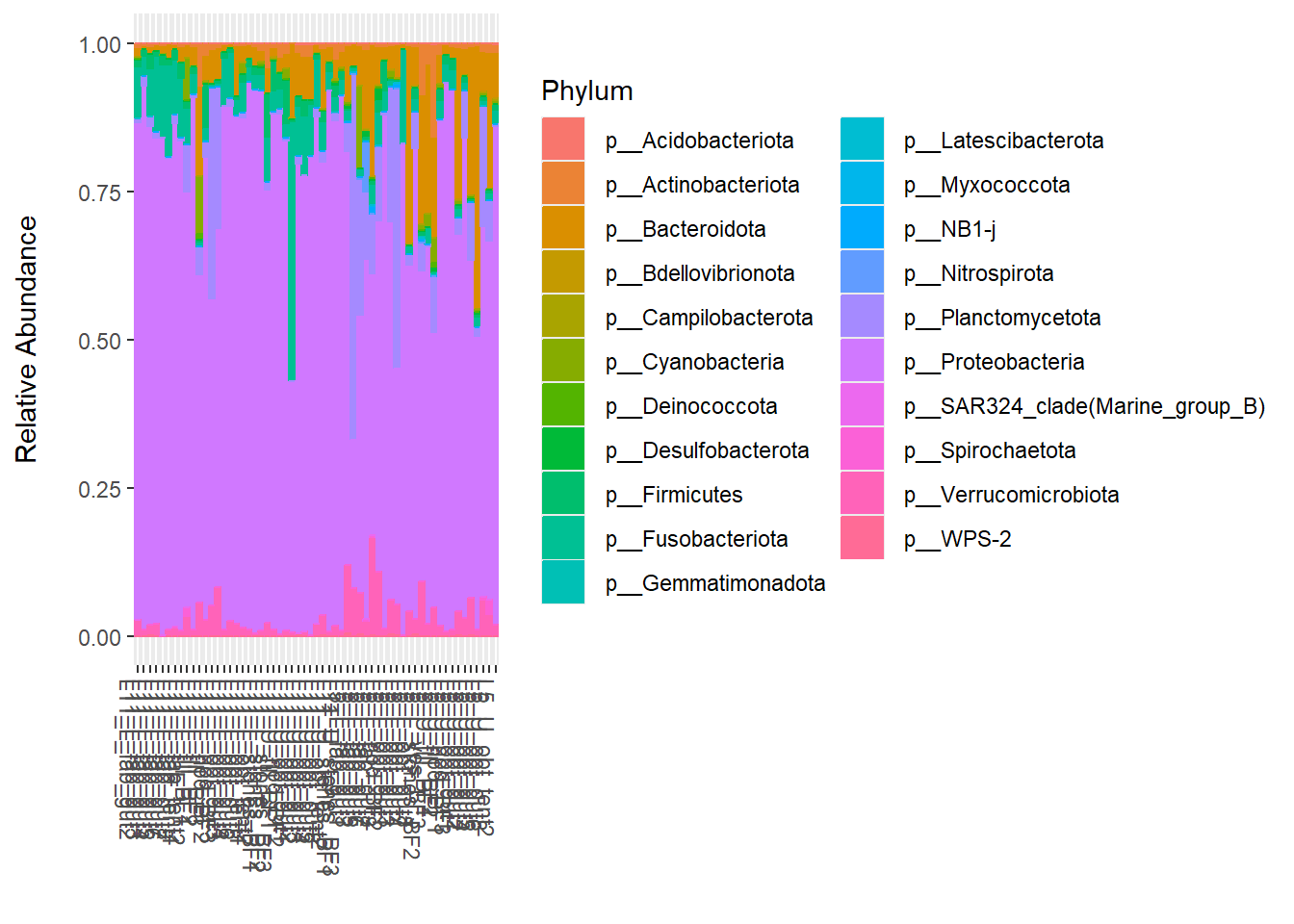
5.1 для всего датасета
5.1.1 встроенное в филосек
5.1.2 специальный пакет для вложенных столбиков
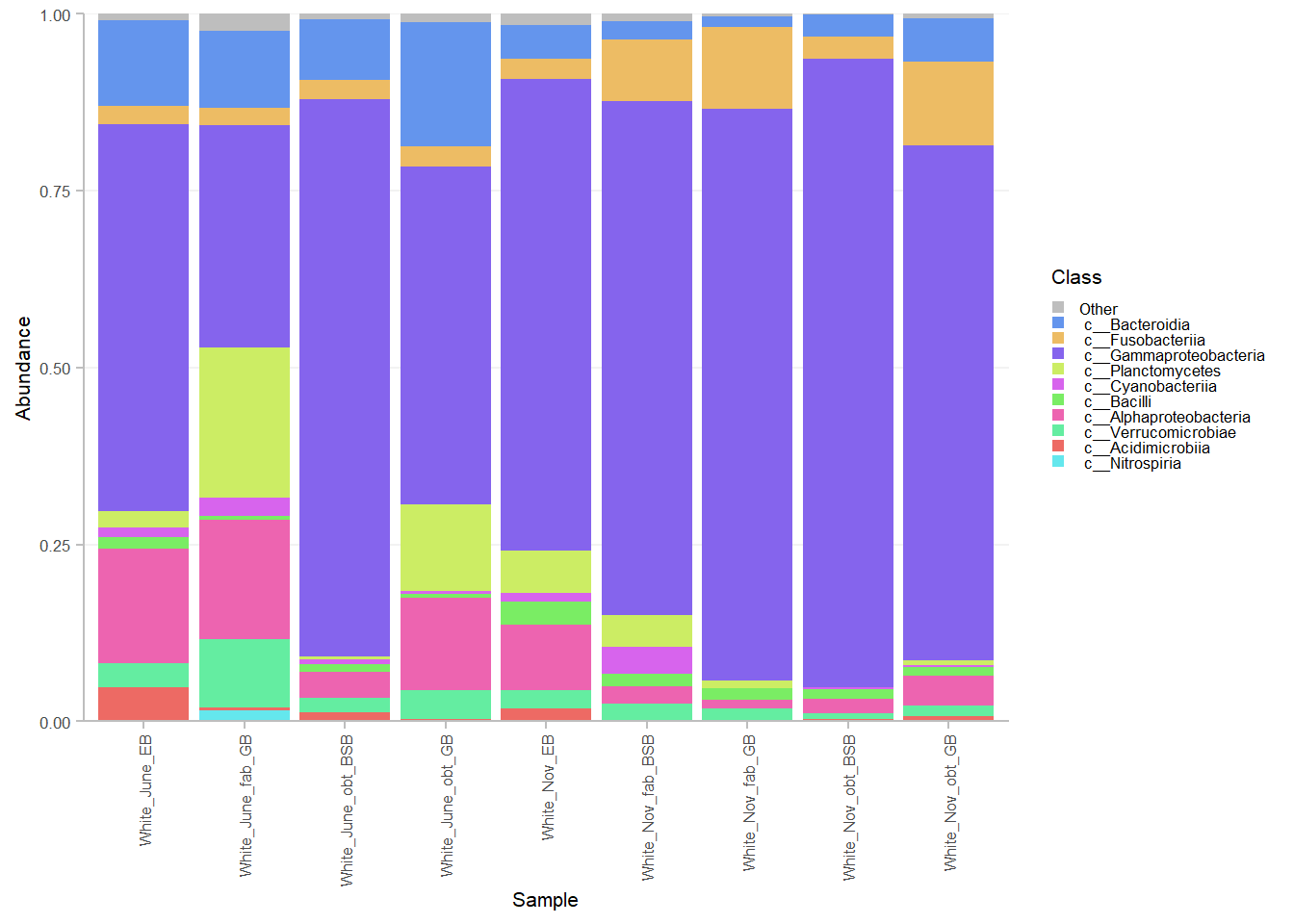
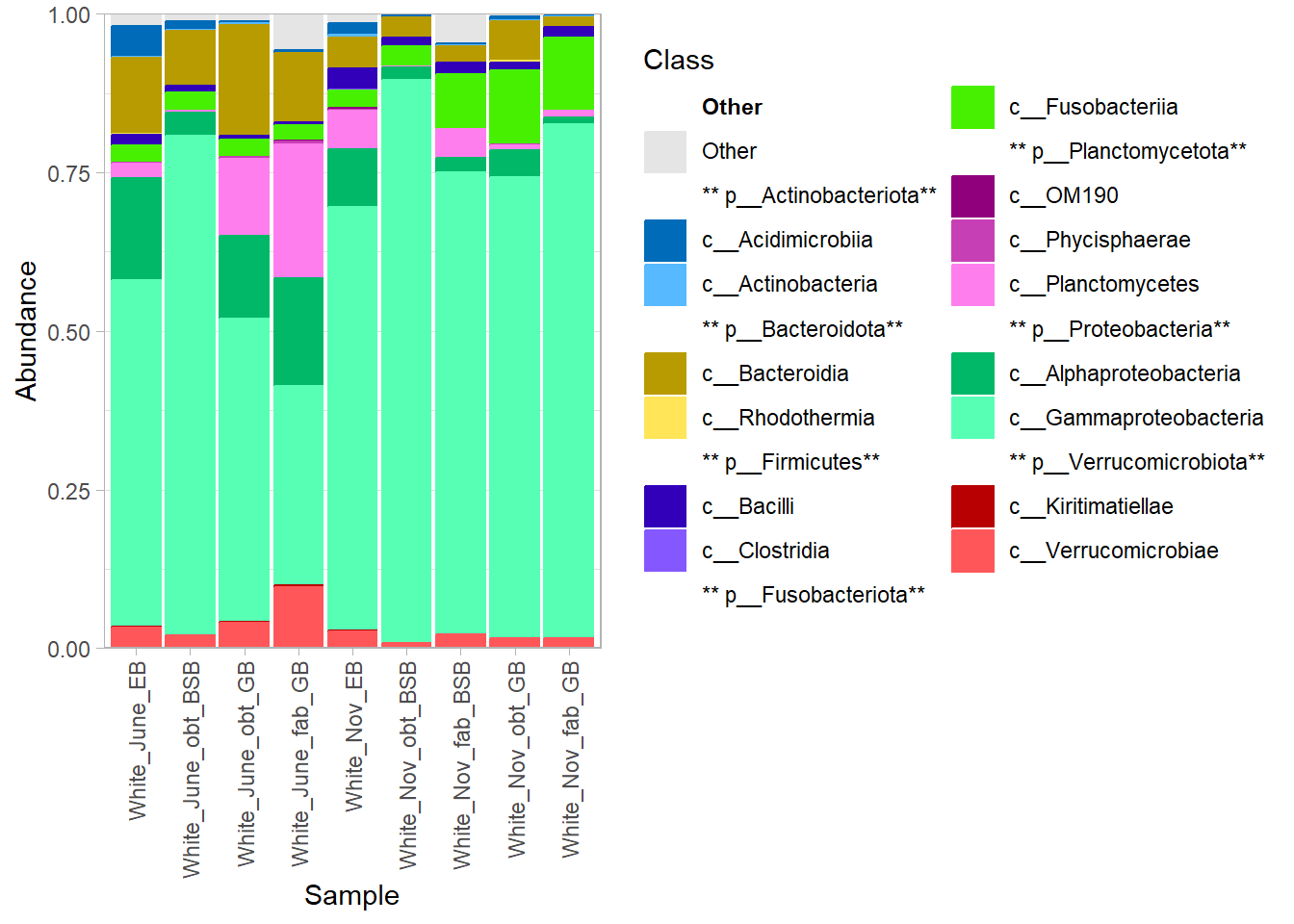
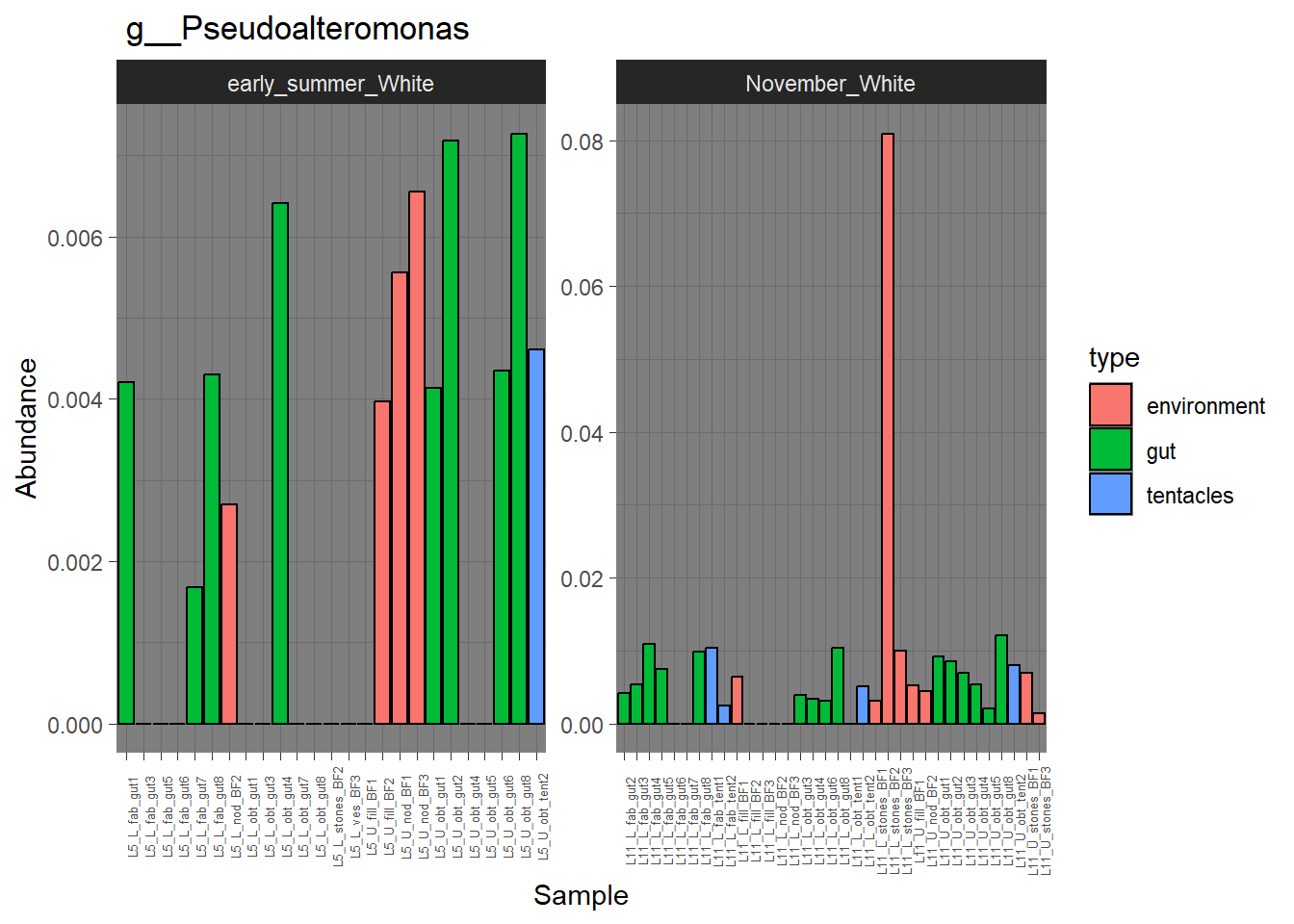
5.2 Барплоты для отдельных видов
5.2.1 Pseudoalteromonas
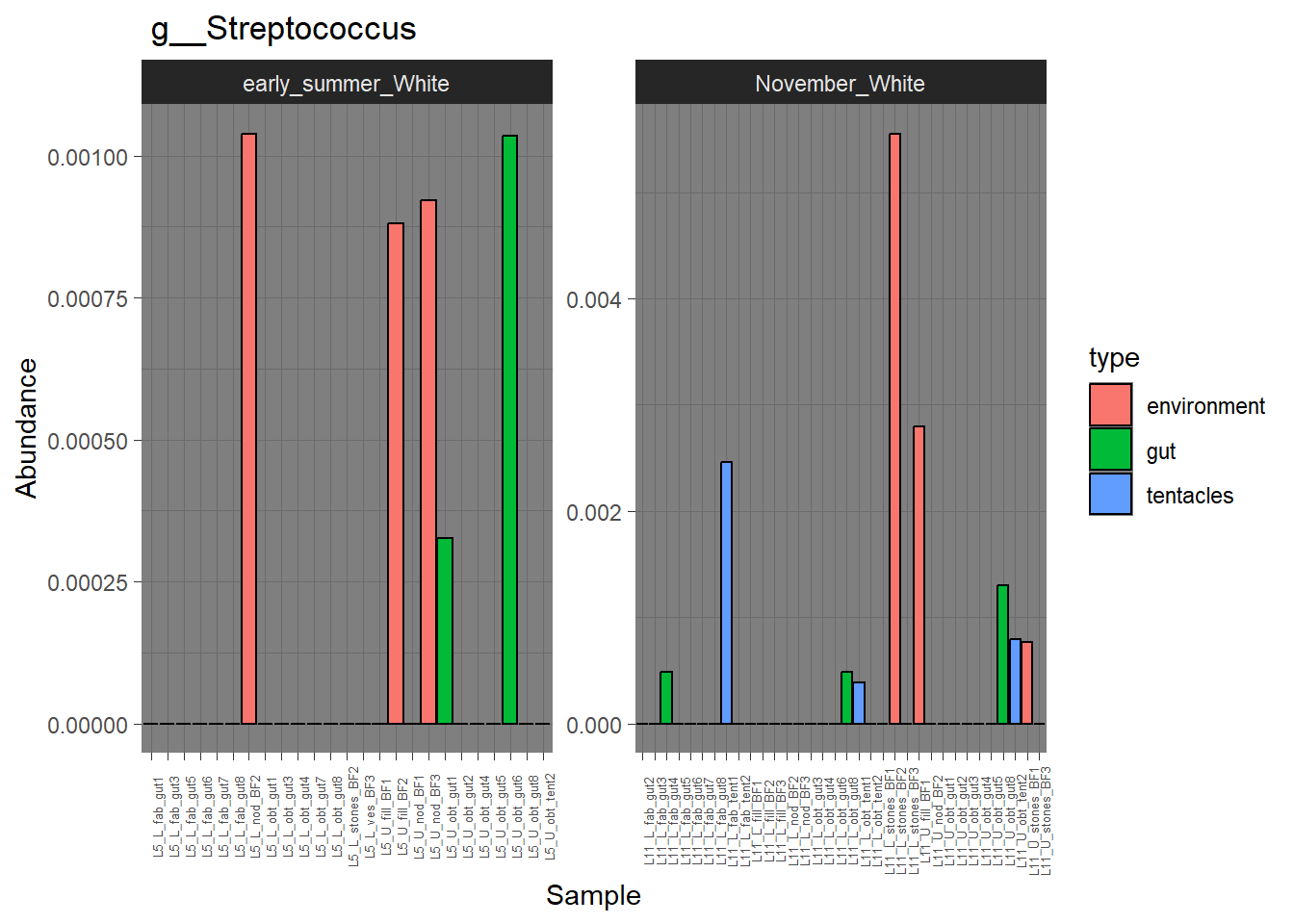
5.2.2 Streptococcus
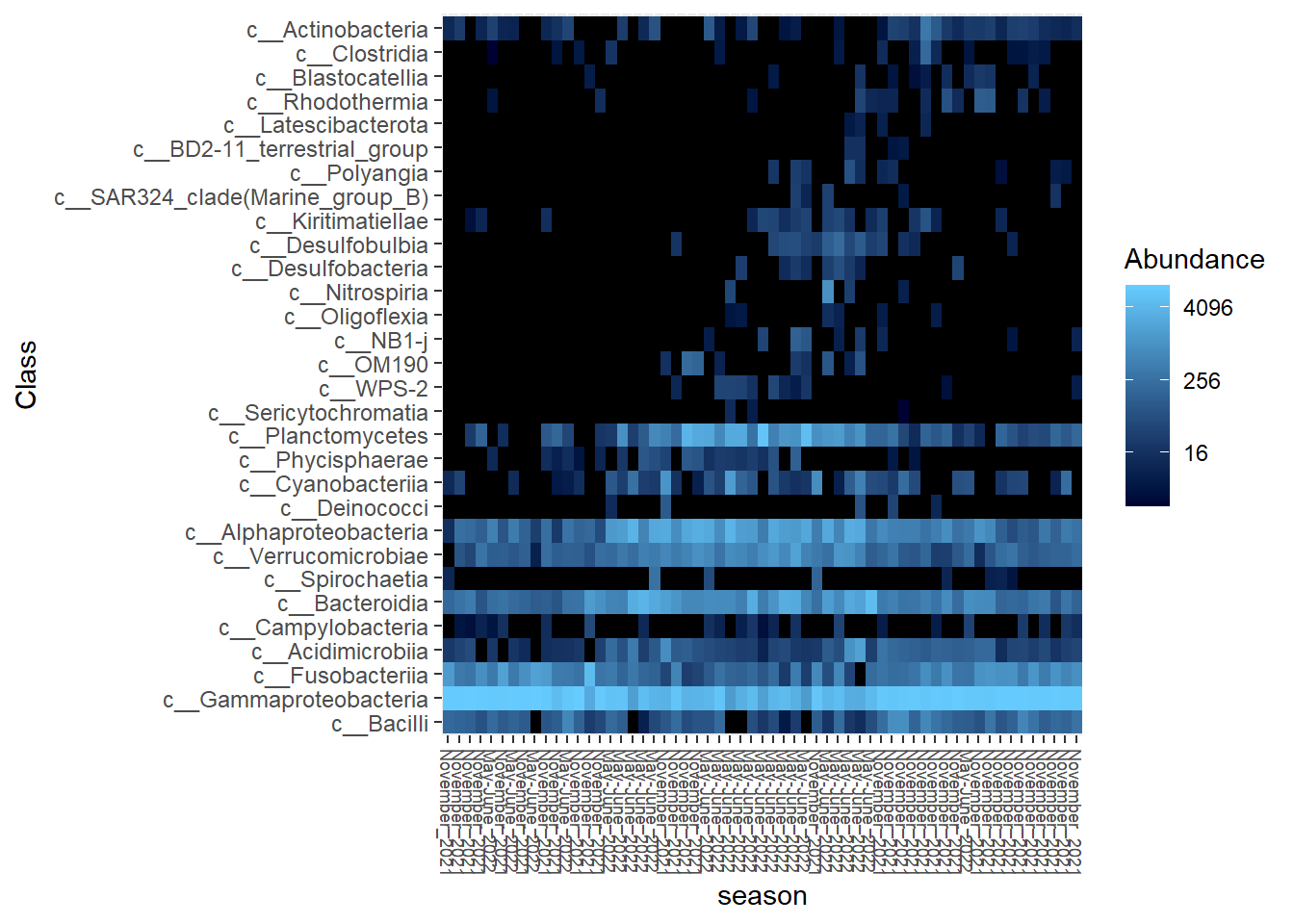
6 Бонусный пример хитмапа