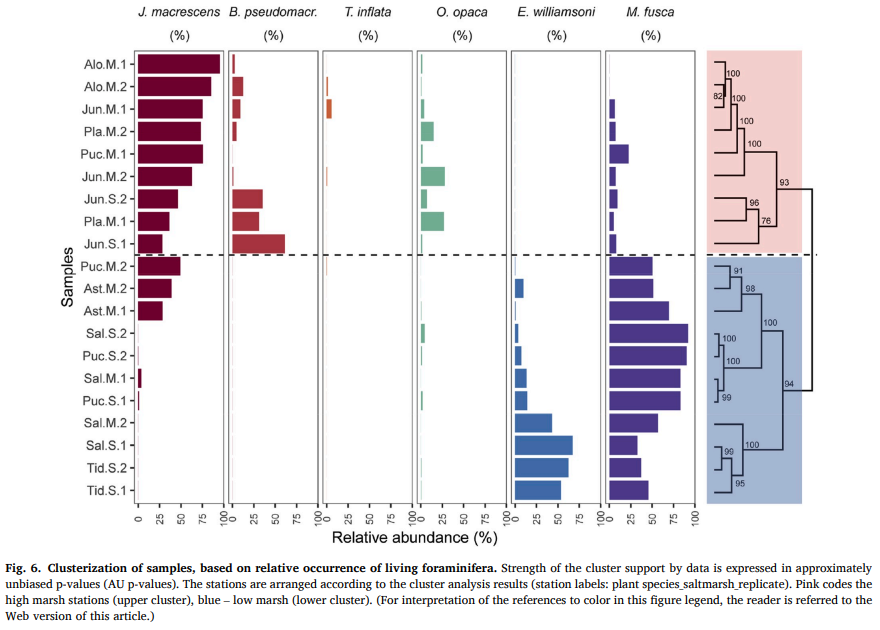
class: middle, left, inverse, title-slide .title[ # Кластерный анализ ] .subtitle[ ## Анализ и визуализация многомерных данных с использованием R ] .author[ ### Марина Варфоломеева ] .author[ ### Вадим Хайтов ] .author[ ### Анастасия Лянгузова ] --- ## Кластерный анализ - Методы построения деревьев - Методы кластеризации на основании расстояний - Примеры для демонстрации и для заданий - Кластерный анализ в R - Качество кластеризации: - кофенетическая корреляция - ширина силуэта - поддержка ветвей - Сопоставление деревьев: танглграммы - Неиерархические методы кластеризации: - K-means - C-means - DBSCAN --- ### Вы сможете - Выбирать подходящий метод агрегации (алгоритм кластеризации) - Строить дендрограммы - Оценивать качество кластеризации (кофенетическая корреляция, ширина силуэта, поддержка ветвей) - Сопоставлять дендрограммы, полученные разными способами, при помощи танглграмм --- class: middle, center, inverse # Пример: Волки --- ## Пример: Волки Морфометрия черепов у волков в Скалистых горах и в Арктике (Jolicoeur, 1959) 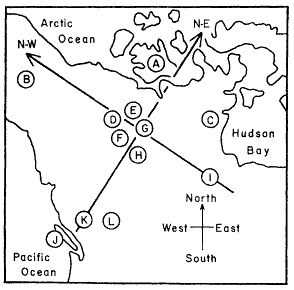 Данные взяты из работы Morrison (1990): - A --- волки из Арктики (10 самцов, 6 самок) - L --- волки из Скалистых гор (6 самцов, 3 самки) ``` r library(candisc) data("Wolves") ``` --- ## Знакомимся с данными ``` r dim(Wolves) ``` ``` [1] 25 12 ``` ``` r colnames(Wolves) ``` ``` [1] "group" "location" "sex" "x1" "x2" "x3" "x4" [8] "x5" "x6" "x7" "x8" "x9" ``` ``` r head(rownames(Wolves)) ``` ``` [1] "rmm1" "rmm2" "rmm3" "rmm4" "rmm5" "rmm6" ``` ``` r any(is.na(Wolves)) ``` ``` [1] FALSE ``` ``` r table(Wolves$group) ``` ``` ar:f ar:m rm:f rm:m 6 10 3 6 ``` --- ## Задание 1. Постройте ординацию nMDS данных. 1. Оцените качество ординации. 1. Обоснуйте выбор коэффициента. 1. Раскрасьте точки на ординации волков в зависимости от географического происхождения (`group`). --- ## Решение .scroll-box-20[ ``` r library(vegan) library(ggplot2); theme_set(theme_bw(base_size = 16)) st_w <- scale(Wolves[, 4:ncol(Wolves)]) ## стандартизируем ord_w <- metaMDS(comm = st_w, distance = "euclidean", autotransform = FALSE) dfr_w <- data.frame(ord_w$points, Group = Wolves$group) gg_w <- ggplot(dfr_w, aes(x = MDS1, y = MDS2)) + geom_point(aes(colour = Group)) + scale_color_manual(labels = c("Самки из Арктики", "Самцы из Арктики", "Самки из Скалистых гор", "Самцы из скалистых гор"), values = c("red", "green", "blue", "purple")) + labs(colour = "Группы") ``` ``` Run 0 stress 0.101 Run 1 stress 0.1492 Run 2 stress 0.1244 Run 3 stress 0.101 ... New best solution ... Procrustes: rmse 0.000005603 max resid 0.00001869 ... Similar to previous best Run 4 stress 0.1357 Run 5 stress 0.1244 Run 6 stress 0.101 ... Procrustes: rmse 0.000004521 max resid 0.00001452 ... Similar to previous best Run 7 stress 0.101 ... Procrustes: rmse 0.0000125 max resid 0.00004729 ... Similar to previous best Run 8 stress 0.101 ... Procrustes: rmse 0.000006085 max resid 0.00002023 ... Similar to previous best Run 9 stress 0.1492 Run 10 stress 0.101 ... Procrustes: rmse 0.000002464 max resid 0.000007485 ... Similar to previous best Run 11 stress 0.1382 Run 12 stress 0.1403 Run 13 stress 0.1275 Run 14 stress 0.1415 Run 15 stress 0.1389 Run 16 stress 0.1281 Run 17 stress 0.128 Run 18 stress 0.1511 Run 19 stress 0.101 ... Procrustes: rmse 0.000001249 max resid 0.000003076 ... Similar to previous best Run 20 stress 0.1412 *** Best solution repeated 6 times ``` ] --- ## Решение 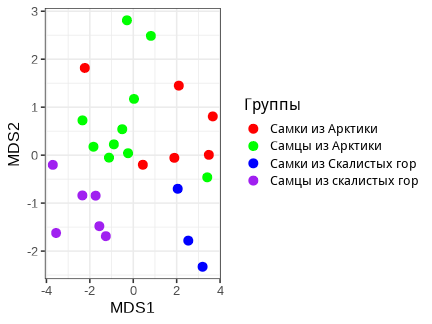<!-- --> --- ## Методы кластеризации .pull-left[ ### Иерархические методы - методы построения деревьев (о них следующие слайды) ] .pull-right[ ### Неиерархические методы - метод K-средних (K-means clustering) - метод нечёткой кластеризации C-средних (C-means clustering, fuzzy clustering) - Основанная на плотности пространственная кластеризация для приложений с шумами (Density-based spatial clustering of applications with noise, DBSCAN) ] --- ## Какие бывают методы построения деревьев? ### Методы кластеризации на основании расстояний (о них сегодня) - Метод ближайшего соседа - Метод отдалённого соседа - Метод среднегруппового расстояния - Метод Варда - и т.д. и т.п. ### Методы кластеризации на основании признаков - Метод максимальной бережливости - Метод максимального правдоподобия ### И это еще далеко не всё! --- class: middle, center, inverse # Методы кластеризации на основании расстояний --- ## Этапы кластеризации .pull-left[ <div class="DiagrammeR html-widget html-fill-item" id="htmlwidget-1238a4c60bc70b0b6f4e" style="width:432px;height:540px;"></div> <script type="application/json" data-for="htmlwidget-1238a4c60bc70b0b6f4e">{"x":{"diagram":"graph TD; \n A(Набор признаков)-->B(Матрица расстояний или сходств); \n B-->C(Группировка объектов);\n C-->D(Отношения между кластерами); D-->E(Поправки); E-->A;\n style A fill:#C6FFE7,stroke:#C6FFE7;\n style B fill:#C6FFE7,stroke:#C6FFE7;\n style C fill:#C6FFE7,stroke:#C6FFE7;\n style D fill:#C6FFE7,stroke:#C6FFE7;\n style E fill:#C6FFE7,stroke:#C6FFE7;\n style A color:#C6FFE7,stroke:#C6FFE7;\n "},"evals":[],"jsHooks":[]}</script> ] .pull-right[ Результат кластеризации зависит от - выбора признаков - коэффициента сходства-различия - от алгоритма кластеризации ] --- ## Методы кластеризации 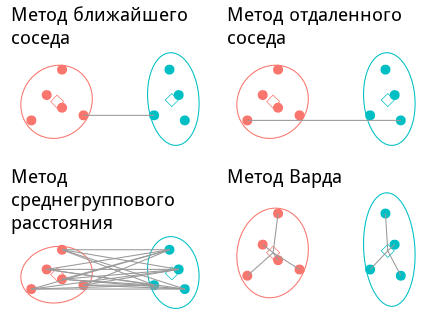<!-- --> --- ## Метод ближайшего соседа .pull-left-66[ .content-box-green[ __= nearest neighbour = single linkage__ - к кластеру присоединяется ближайший к нему кластер/объект - кластеры объединяются в один на расстоянии, которое равно расстоянию между ближайшими объектами этих кластеров ] ] .pull-right-33[ <!-- --> ] ### Особенности - Может быть сложно интерпретировать, если нужны группы - объекты на дендрограмме часто не образуют четко разделенных групп - часто получаются цепочки кластеров (объекты присоединяются как бы по одному) - Хорош для выявления градиентов --- ## Как работает метод ближайшего соседа <!-- --> --- ## Как работает метод ближайшего соседа <!-- --> --- ## Метод отдаленного соседа .pull-left-60[ .content-box-green[ __= furthest neighbour = complete linkage__ - к кластеру присоединяется отдаленный кластер/объект - кластеры объединяются в один на расстоянии, которое равно расстоянию между самыми отдаленными объектами этих кластеров (следствие — чем более крупная группа, тем сложнее к ней присоединиться) ]] .pull-right-25[ <!-- --> ] ### Особенности - На дендрограмме образуется много отдельных некрупных групп - Хорош для поиска дискретных групп в данных --- ## Как работает метод отдаленного соседа <!-- --> --- ## Как работает метод отдаленного соседа <!-- --> --- ## Метод невзвешенного попарного среднего .pull-left-66[ .content-box-green[ __= UPGMA = Unweighted Pair Group Method with Arithmetic mean__ кластеры объединяются в один на расстоянии, которое равно среднему значению всех возможных расстояний между объектами из разных кластеров. ]] .pull-right-33[ <!-- --> ] ### Особенности UPGMA и WUPGMС иногда могут приводить к инверсиям на дендрограммах .pull-left-66[  ] .pull-right-33[ .pull-down[ из Borcard et al., 2011 ]] --- ## Как работает метод среднегруппового расстояния <!-- --> --- ## Как работает метод среднегруппового расстояния <!-- --> --- ## Метод Варда .pull-left-66[.content-box-green[ __= Ward's Minimum Variance Clustering__ объекты объединяются в кластеры так, чтобы внутригрупповая дисперсия расстояний была минимальной. ] <!-- --> ] .pull-left-33[ ### Особенности метод годится и для неевклидовых расстояний несмотря на то, что внутригрупповая дисперсия расстояний рассчитывается так, как будто это евклидовы расстояния.] --- ## Как работает метод Варда <!-- --> --- ## Как работает метод Варда <!-- --> --- class: middle, center, inverse # Кластерный анализ в R --- ## Кластеризация Давайте построим деревья при помощи нескольких алгоритмов кластеризации (по стандартизованным данным, с использованием Евклидова расстояния) и сравним их. ``` r # Пакеты для визуализации кластеризации library(ape) library(dendextend) ``` ``` r # Матрица расстояний d <- dist(x = st_w, method = "euclidean") ``` --- ## (1.0) Метод ближайшего соседа + `base` ``` r hc_single <- hclust(d, method = "single") plot(hc_single) ``` <!-- --> --- ## (1.1) Метод ближайшего соседа + `ape` ``` r ph_single <- as.phylo(hc_single) plot(ph_single, type = "phylogram") axisPhylo() ``` <!-- --> --- ## (1.2) Метод ближайшего соседа + `dendextend` ``` r den_single <- as.dendrogram(hc_single) plot(den_single, horiz = TRUE) ``` <!-- --> --- ## (2.1) Метод отдаленного соседа + `ape` ``` r hc_compl <- hclust(d, method = "complete") ph_compl <- as.phylo(hc_compl) plot(ph_compl, type = "phylogram") axisPhylo() ``` <!-- --> --- ## (2.2)Метод отдаленного соседа + `dendextend` ``` r den_compl <- as.dendrogram(hc_compl) plot(den_compl, horiz = TRUE) ``` <!-- --> --- ## (3.1) Метод невзвешенного попарного среднего (UPGMA) + `ape` ``` r hc_avg <- hclust(d, method = "average") ph_avg <- as.phylo(hc_avg) plot(ph_avg, type = "phylogram") axisPhylo() ``` <!-- --> --- ## (3.2) Метод невзвешенного попарного среднего (UPGMA) + `dendextend` ``` r den_avg <- as.dendrogram(hc_avg) plot(den_avg, horiz = TRUE) ``` <!-- --> --- ## (4.1) Метод Варда + `ape` ``` r hc_w2 <-hclust(d, method = "ward.D2") ph_w2 <- as.phylo(hc_w2) plot(ph_w2, type = "phylogram") axisPhylo() ``` <!-- --> --- ## (4.2) Метод Варда + `dendextend` ``` r den_w2 <- as.dendrogram(hc_w2) plot(den_w2, horiz = TRUE) ``` <!-- --> --- class: middle, center, inverse # Качество кластеризации --- ## Кофенетическая корреляция __Кофенетическое расстояние__ — расстояние между объектами на дендрограмме, т.е. то расстояние, на котором объекты становятся частью одной группы в ходе процесса кластеризации. .content-box-green[ __Кофенетическая корреляция__ — мера качества отображения многомерных данных на дендрограмме. Кофенетическую корреляцию можно рассчитать как Пирсоновскую корреляцию (обычную) между матрицами исходных и кофенетических расстояний между всеми парами объектов. В идеальном случае равна 1. ] <br/> Метод агрегации, который дает наибольшую кофенетическую корреляцию, дает кластеры, лучше всего отражающие исходные данные. Матрицу кофенетических расстояний и кофенетическую корреляцию можно рассчитать при помощи функций из пакета `stats` (`dendextend`) и `ape`, соответственно. --- ## Кофенетическая корреляция в R ``` r # Матрица кофенетических расстояний c_single <- cophenetic(ph_single) # Кофенетическая корреляция = # = корреляция матриц кофенетич. и реальн. расстояний cor(d, as.dist(c_single)) ``` ``` [1] 0.5654 ``` --- ## Задание: Оцените при помощи кофенетической корреляции качество кластеризаций, полученных разными методами. Какой метод дает лучший результат? --- ## Решение: ``` r c_single <- cophenetic(ph_single) cor(d, as.dist(c_single)) ``` ``` [1] 0.5654 ``` ``` r c_compl <- cophenetic(ph_compl) cor(d, as.dist(c_compl)) ``` ``` [1] 0.7058 ``` ``` r c_avg <- cophenetic(ph_avg) cor(d, as.dist(c_avg)) ``` ``` [1] 0.7447 ``` ``` r c_w2 <- cophenetic(ph_w2) cor(d, as.dist(c_w2)) ``` ``` [1] 0.726 ``` --- ## Что можно делать дальше с дендрограммой - Можно выбрать число кластеров: + либо субъективно, на любом выбранном уровне (главное, чтобы кластеры были осмысленными и интерпретируемыми); + либо исходя из распределения расстояний ветвления. - Можно оценить стабильность кластеризации при помощи бутстрепа. --- ## Ширина силуэта .content-box-green[ Ширина силуэта `\(s_i\)` --- мера степени принадлежности объекта `\(i\)` к кластеру ] `$$s_i = \frac {\color{purple}{\bar{d}_{i~to~nearest~cluster}} - \color{green}{\bar{d}_{i~within}}} {max\{\color{purple}{\bar{d}_{i~to~nearest~cluster}}, \color{green}{\bar{d}_{i~within}}\}}$$` `\(s_i\)` --- сравнивает между собой средние расстояния от данного объекта: - `\(\color{green} {\bar{d}_{i~within}}\)` --- до других объектов из того же кластера - `\(\color{purple} {\bar{d}_{i~to~nearest~cluster}}\)` --- до ближайшего кластера `\(-1 \le s_i \le 1\)` --- чем больше `\(s_i\)`, тем "лучше" объект принадлежит кластеру. - Средняя ширина силуэта для всех объектов из кластера --- оценивает, насколько "тесно" сгруппированы объекты. - Средняя ширина силуэта по всем данным --- оценивает общее качество классификации. - Чем больше к 1, тем лучше. Если меньше 0.25, то можно сказать, что нет структуры. --- ## Как рассчитывается ширина силуэта Оценим ширину силуэта для 3 кластеров ``` r library(cluster) avg3 <- cutree(tree = hc_avg, k = 3) # делим дерево на нужное количество кластеров plot(silhouette(x = avg3, dist = d), cex.names = 0.6) ``` <!-- --> --- ## Бутстреп .content-box-green[ __Бутстреп__ --- один из методов оценки значимости полученных результатов; повторная выборка из имеющихся данных. В такой выборке элементы могут повторяться.] ### Алгоритм - Имеющиеся данные делим на группы одинакового размера: какие-то элементы могут отсутствовать, а какие-то --- повторяться несколько раз. - На основе полученных данных строим дендрограмму. - Повторяем всё много раз. --- ## Бутстреп поддержка ветвей > "An approximately unbiased test of phylogenetic tree selection." > > --- Shimodaria, 2002 Этот тест использует специальный вариант бутстрепа --- multiscale bootstrap. Мы не просто многократно берем бутстреп-выборки и оцениваем для них вероятность получения топологий (BP p-value), эти выборки еще и будут с разным числом объектов. По изменению BP при разных объемах выборки можно вычислить AU (approximately unbiased p-value). ``` r library(pvclust) set.seed(389) # итераций должно быть 1000 и больше, здесь мало для скорости cl_boot <- pvclust(t(st_w), method.hclust = "average", nboot = 100, method.dist = "euclidean", parallel = TRUE, iseed = 42) ``` ``` Creating a temporary cluster...done: socket cluster with 11 nodes on host 'localhost' Multiscale bootstrap... Done. ``` Обратите внимание на число итераций: `nboot = 100` --- это очень мало. На самом деле нужно 10000 или больше. --- ## Дерево с величинами поддержки AU --- approximately unbiased p-values (красный), BP --- bootstrap p-values (зеленый) ``` r plot(cl_boot) pvrect(cl_boot) # достоверные ветвления ``` <!-- --> --- ## Для диагностики качества оценок AU График стандартных ошибок для AU p-value нужен, чтобы оценить точность оценки самих AU. Чем больше было бутстреп-итераций, тем точнее будет оценка AU. ``` r seplot(cl_boot) # print(cl_boot) # все значения ``` <!-- --> --- class: middle, center, inverse # Сопоставление деревьев: Танглграммы --- ## Танглграмма Два дерева (с непохожим ветвлением) выравнивают, вращая случайным образом ветви вокруг оснований. Итеративный алгоритм. Картина каждый раз разная. ``` r set.seed(395) untang_w <- untangle_step_rotate_2side(den_compl, den_w2, print_times = F) # танглграмма tanglegram(untang_w[[1]], untang_w[[2]], highlight_distinct_edges = FALSE, common_subtrees_color_lines = F, main = "Tanglegram", main_left = "Left tree", main_right = "Right tree", columns_width = c(8, 1, 8), margin_top = 3.2, margin_bottom = 2.5, margin_inner = 4, margin_outer = 0.5, lwd = 1.2, edge.lwd = 1.2, lab.cex = 1.5, cex_main = 2) ``` --- ## Танглграмма <!-- --> --- ## Задание Постройте танглграмму из дендрограмм, полученных методом ближайшего соседа и методом Варда. --- ## Раскраска дендрограмм ### Вручную ``` r # Произвольные цвета радуги cols <- rainbow(30) den_avg_manual <- color_labels(dend = den_avg, col = cols) plot(den_avg_manual, horiz = TRUE) ``` <!-- --> --- ## Раскраска дендрограмм ### С помощью функции .pull-left[ ``` r # Функция для превращения лейблов в цвета # (группы определяются по `n_chars` первых букв в лейбле) get_colours <- function(dend, n_chars, palette = "Dark2"){ labs <- get_leaves_attr(dend, "label") group <- substr(labs, start = 0, stop = n_chars) group <- factor(group) cols <- brewer.pal(length(levels(group)), name = palette)[group] return(cols) } ``` ] .pull-right[ ``` r library(RColorBrewer) cols <- get_colours(dend = den_avg, n_chars = 3) den_avg_c <- color_labels(dend = den_avg, col = cols) plot(den_avg_c, horiz = TRUE) ``` <!-- --> ] --- class: middle, center, inverse ## Неиерархические методы кластеризации --- ## Метод K-средних (K-means) .pull-left[ <!-- --> ] .pull-right[ В отличие от иерархических методов кластеризации K-means будет искать то количество кластеров (k), которое вы ему зададите. Каждое наблюдение принадлежит кластеру с ближайшим значением среднего числа (центроида); помимо этого K-means кластеризация минимизирует разброс значений внутри каждого из кластера. Используется в машинном обучении, в том числе, например, для цветовой редуцкии изображений. ] --- ## Алгоритм K-means .pull-left[ ### График наблюдений <!-- --> Здесь как будто бы выделяются 3 кластера, поэтому возьмём k = 3. Что же будет делать алгоритм? ] -- .pull-right[ ### 1. Выбираются случайным образом 3 точки на графике --- кластерные центроиды Например, так: <!-- --> ] --- ## Алгоритм K-means .pull-left[ ### 2. Измеряется Евклидово расстояние между каждой точкой и центроидом При этом каждая точка приписывается к ближайшему кластеру. <!-- --> ] .pull-right[ ### 3. Расситываются центроиды для каждого кластера <!-- --> ] --- ## 4. Расчёт расстояний от каждой точки до нового центроида .pull-left[ Также оценивается разброс внутри каждого кластера. <!-- --> ] .pull-right[ Разброс считается как сумма квадратов расстояний между отдельными наблюдениями и центроидом. $$ \sum_{i=1}^{n}(x_i - \overline{x})$$ ] --- ## 5. Повторяем всё многократно до тех пор, пока разброс не станет минимальным Кластеры с минимальным разбросом --- финальные. <!-- --> --- ## Поиск локального минимума  .tiny[ Из Legendre, Legendre, 2012 ] Кратко алгоритм можно свести к поиску общего минимума среди локальных (вне зависимости от начальной конфигурации). --- ## K-means в R Если в данных много нулей, их обязательно нужно стандартизовать (что мы уже делали). K-means кластеризацию в R делает функция `kmeans` (пакет `stats`), ей нужно передать аргументы `centers` (количество кластеров) и `nstart` (количество случайных итераций). ``` r set.seed(333) w_kmeans <- kmeans(st_w, centers = 3, nstart = 100) ``` Полученные результаты можно сравнить с тем, что нам дала иерархическая кластеризация (UPGMA) --- оценка ширины силуэта. ``` r table(avg3, w_kmeans$cluster) ``` ``` avg3 1 2 3 1 6 0 9 2 0 7 0 3 0 1 2 ``` --- ## Как выбрать нужное количество кластеров? Два популярных критерия: - Индекс Калински-Харабаза (Calinski–Harabasz index): F-статистика, сравнивающая меж- и внутригрупповую сумму квадратов. Если группы одинаковой величины. - Индекс простой структуры (Simple Structure Index): оценивает влияние на интепретабельность полученной кластеризации. Если группы разной величины. --- ## Как выбрать нужное количество кластеров? Функция `cascadeKM` из пакета `vegan`. По сути функция-обёртка, которая проводит кластеризацию с разным заданным количеством кластеров. ``` r w_cascade <- cascadeKM(st_w, inf.gr = 2, sup.gr = 10, iter = 100, criterion = 'calinski') ``` - `inf.gr` --- начальное количество кластеров, - `sup.gr` --- максимальное количество кластеров, - `iter` --- количество итераций для каждой кластеризации, - `criterion` --- индекс: `calinski` или `ssi`. --- ## Визуализация результатов множественной кластеризации ``` r plot(w_cascade, sortg = TRUE) # чтобы объекты, относящиеся к одному кластеру, рисовались вместе ``` <!-- --> Советует 2 кластера! --- ## Визуализация k-means Делается с помощью функции `fviz_cluster` из пакета `factoextra`. .pull-left[ ``` r library(factoextra) fviz_cluster(w_kmeans, data = st_w, ggtheme = theme_bw()) ``` <!-- --> ] .pull-right[ ``` r w_2k <- kmeans(st_w, centers = 2, nstart = 100) fviz_cluster(w_2k, data = st_w, ggtheme = theme_bw()) ``` <!-- --> ] --- ## Оценка качества кластеризации: ширина силуэта .pull-left[ ``` r plot(silhouette(w_kmeans$cluster, d)) ``` <!-- --> ] .pull-right[ ``` r plot(silhouette(w_2k$cluster, d)) ``` <!-- --> ] --- ## Density-based spatial clustering of applications with noise (DBSCAN) .pull-left[ Основанная на плотности пространственная кластеризация для приложений с шумами --- метод, более подходящий для "вложенных" кластеров. Основан на распределении плотности точек. <!-- --> ] .pull-right[ Работает с данными, с которыми другие методы кластеризации не могут справиться (K-means в примере). ``` r set.seed(123) circle_kmeans <- kmeans(multi, centers = 2, nstart = 20) my_col_circle <- c("#2E9FDF", "#E7B800") fviz_cluster(circle_kmeans, data = multi, palette = my_col_circle) ``` <!-- --> ] --- ## Принцип работы DBSCAN Кластеры выбираются на основе плотности расположения точек. В результате в единый кластер объединяются близко расположенные друг к другу точки. Задаваемые параметры: - радиус расстояния, на котором должны рассматриваться близлежащие точки (`eps`) - минимальное количество точек, которые расположены в круге этого радиуса (`minPts`) Сore points --- точки, от которых можем присоединять в кластер новые точки и рядом с которыми расположено `minPts` количество точек. Пограничные точки --- те, на которых кластер заканчивается. Остальные точки считаются шумом и выбросами. --- ## DBSCAN в R Провести такую кластеризацию можно с помощью функции `dbscan` из пакета `dbscan`. <!-- --> --- ## Epsilon --- выбираем расстояние для радиуса **График k-расстояний (k-distance plot)** 1. Вычисляется среднее значение расстояний каждой точки до ее k ближайших соседей. 2. k-расстояния отображаются в порядке возрастания. Если на графике есть "колено" --- значительный перегиб, будет легко найти нужное значение радиуса. .pull-left[ ``` r kNNdistplot(multi, k = 5) abline(h = 0.23, lty = 2) ``` <!-- --> ] .pull-right[ ``` r circle_dbscan <- dbscan(multi, 0.23, 5) fviz_cluster(circle_dbscan, data = multi, palette = my_col_circle) ``` <!-- --> ] --- ## Задание 4 Кластеризуйте данные по волкам, используя DBSCAN-алгоритм. --- ## Примерное решение .pull-left[ ``` r kNNdistplot(st_w, k = 4) abline(h = 2.4, lty = 2) ``` <!-- --> ] .pull-right[ ``` r w_dbscan <- dbscan(st_w, 2.4, 4) fviz_cluster(w_dbscan, data = st_w) ``` <!-- --> ] --- ## Метод нечёткой кластеризации C-средних (C-means, fuzzy clustering) При данном подходе объект необязательно принадлежит одному кластеру. Каждому объекту присваивается вероятность принадлежности к кластеру (membership value). В сумме все значения принадлежности к кластеру дают 1 для каждого из объектов. --- ## Алгоритм C-means 1. Каждому объекту приписывается случайное значение принадлежности к кластеру. 2. Рассчитываются центроиды для каждого кластера: `$$\hat{v}_{i} = \frac{\sum^{N}_{k=1}(\hat{u}_{ik})^{m}y_k}{\sum^{N}_{k=1}(\hat{u}_{ik})^m}$$` где `\(\hat{u}\)` --- значение принадлежности к кластеру, `\(m\)` --- параметр размытости (fuzziness), равный обычно 2, `\(y_k\)` --- конкретный объект, N --- количество объектов. --- ## Алгоритм C-means <ol start="3"> <li> Расчёт расстояния от каждой точки до центроида. <li> Обновление значений принадлежности к кластерам. </ol> `$$\hat{u}_{ik} = (\sum^{c}_{j=1}(\frac{\hat{d}_{ik}}{\hat{d}_{jk}})^{\frac{2}{m-1}})^{-1}$$` где `\(d_{ki}\)` --- расстояние от точки до центроида. <ol start="5"> <li> Повторить шаги с 2 по 4, пока не будут получены постоянные значения принадлежности к кластеру. </ol> --- ## Кластеризация C-means в R Есть несколько функций из разных пакетов, реализующих кластеризацию С-средних (например `fanny` из `cluster`, `cmeans` из `e1071`, `fcm` из `ppclust` и т.д.). Мы воспользуемся функцией `fanny` из пакета `cluster`. Функция funny принимает как исходные данные, так и матрицу расстояний. ``` r w_cmeans <- fanny(d, k = 4, memb.exp = 2) summary(w_cmeans) ``` ``` Fuzzy Clustering object of class 'fanny' : m.ship.expon. 2 objective 12.07 tolerance 1e-15 iterations 85 converged 1 maxit 500 n 25 Membership coefficients (in %, rounded): [,1] [,2] [,3] [,4] rmm1 28 17 28 28 rmm2 28 16 28 28 rmm3 28 16 28 28 rmm4 29 14 29 29 rmm5 29 14 29 29 rmm6 28 17 28 28 rmf1 20 39 20 20 rmf2 21 37 21 21 rmf3 22 35 22 22 arm1 25 24 25 25 arm2 28 17 28 28 arm3 27 18 27 27 arm4 28 16 28 28 arm5 28 15 28 28 arm6 29 14 29 29 arm7 29 14 29 29 arm8 26 21 26 26 arm9 26 21 26 26 arm10 21 38 21 21 arf1 21 36 21 21 arf2 20 40 20 20 arf3 25 24 25 25 arf4 22 33 22 22 arf5 27 18 27 27 arf6 21 38 21 21 Fuzzyness coefficients: dunn_coeff normalized 0.26251 0.01667 Closest hard clustering: rmm1 rmm2 rmm3 rmm4 rmm5 rmm6 rmf1 rmf2 rmf3 arm1 arm2 arm3 arm4 arm5 1 1 1 1 1 1 2 2 2 3 3 3 1 1 arm6 arm7 arm8 arm9 arm10 arf1 arf2 arf3 arf4 arf5 arf6 1 1 3 3 2 2 2 3 2 1 2 k_crisp (= 3) < k !! Silhouette plot information: cluster neighbor sil_width rmm4 1 3 0.364448 rmm2 1 3 0.354519 rmm3 1 3 0.348120 rmm5 1 3 0.312708 rmm1 1 3 0.290835 arm5 1 3 0.209294 arm6 1 3 0.190407 rmm6 1 3 0.137320 arf5 1 3 0.042882 arm7 1 3 -0.001203 arm4 1 3 -0.005208 arf2 2 3 0.391057 rmf2 2 3 0.347725 arm10 2 3 0.343759 rmf3 2 3 0.341740 arf6 2 3 0.312072 rmf1 2 3 0.268000 arf1 2 3 0.200487 arf4 2 3 -0.049651 arm1 3 2 0.259831 arm8 3 1 0.233975 arm9 3 1 0.192208 arm3 3 1 0.132853 arf3 3 2 0.083143 arm2 3 1 0.054878 Average silhouette width per cluster: [1] 0.2040 0.2694 0.1595 0.2060 Average silhouette width of total data set: [1] 0.2142 Available components: [1] "membership" "coeff" "memb.exp" "clustering" "k.crisp" [6] "objective" "convergence" "diss" "call" "silinfo" ``` --- ## Оценка качества кластеризации: ширина силуэта ``` r plot(silhouette(w_cmeans), cex.names = 0.6) ``` <!-- --> -- Получилась не очень качественная кластеризация. --- ## Задание 5 Попробуйте оценить ширину силуэта для C-means кластеризации с более подходящим числом кластеров. --- ## Решение ``` r w_2means <- fanny(d, k = 2, memb.exp = 2) plot(silhouette(w_2means), cex.names = 0.6) ``` <!-- --> --- ## Визуализация кластеризации Для визуализации возьмём нашу исходную ординацию nMDS. ``` r library(scatterpie) w_clust <- cbind(dfr_w, w_2means$membership) ggplot() + geom_scatterpie(data = w_clust, aes(x = MDS1, y = MDS2), cols = c("1", "2")) ``` <!-- --> --- ## Take-home messages - Результат кластеризации зависит не только от выбора коэффициента, но и от выбора алгоритма. - Качество кластеризации можно оценить разными способами. - Кластеризации, полученные разными методами, можно сравнить на танглграммах. --- ## Дополнительные ресурсы - Borcard, D., Gillet, F., Legendre, P., 2011. Numerical ecology with R. Springer. - Legendre, P., Legendre, L., 2012. Numerical ecology. Elsevier. - Quinn, G.G.P., Keough, M.J., 2002. Experimental design and data analysis for biologists. Cambridge University Press. --- ## И еще ресурсы Как работает UPGMA можно посмотреть здесь: - http://www.southampton.ac.uk/~re1u06/teaching/upgma/ Как считать поддержку ветвей (пакет + статья): - pvclust: An R package for hierarchical clustering with p-values [WWW Document], n.d. URL http://www.sigmath.es.osaka-u.ac.jp/shimo-lab/prog/pvclust/ (accessed 11.7.14). Для анализа молекулярных данных: - Paradis, E., 2011. Analysis of Phylogenetics and Evolution with R. Springer. Статья про C-means кластеризацию: - Bezdek et al., 1984. FCM: The Fuzzy c-Means Clustering Algorithm. *Computer & Geosciences, 10: 2-3*, 191-203. --- ## Данные для самостоятельной работы ### Фораминиферы маршей Белого моря (Golikova et al. 2020)  --- ## Фораминиферы маршей Белого моря  --- ## Фораминиферы маршей Белого моря .pull-left[  ] .pull-right[ Plate 1. 1. _Balticammina pseudomacrescens_. 2. _Ammotium salsum_. 3. _Jadammina macrescens_. 4. _Trochammina inflata_. 5. _Ammobaculites balkwilli?_ 6. _Miliammina fusca_. 7. _Ovammina opaca_. 8. _Elphidium albiumbilicatum_. 9. _Elphidium williamsoni_. Scale bar 500 μm. ] --- ## Задание Проанализируйте данные об относительных обилиях фораминифер в пробах на Белом море. - Выберите и обоснуйте трансформацию данных и расстояние. - Постройте ординацию nMDS по относительным обилиям фораминифер: - цвет значков --- растение-доминант, - форма значков --- точка сбора. - Постройте дендрограмму проб по сходству относительных обилий фораминифер. - оцените при помощи кофенетической корреляции, какой метод аггрегации лучше, - Постройте визуализацию для методов неиерархической кластеризации - Опишите получившиеся кластеры при помощи различных параметров: - ширина силуэта - бутстреп-поддержка ветвлений --- ## Фораминиферы маршей Белого моря  --- ## Фораминиферы маршей Белого моря