class: middle, left, inverse, title-slide .title[ # Описание, проверка значимости и валидности линейных моделей ] .subtitle[ ## Линейные модели… ] .author[ ### Марина Варфоломеева, Вадим Хайтов, Анастасия Лянгузова ] .date[ ### Осень 2025 ] --- # Описание и проверка значимости линейных моделей ## Вы сможете - Подобрать линейную модель зависимости переменной-отклика от одного предиктора - Протестировать значимость линейной модели в целом и значимость отдельных ее коэффициентов при помощи t и F критериев - Проверить условия применимости линейной регрессии при помощи графиков --- class: middle, center, inverse # Вспомним пример из прошлой лекции --- ## Пример: IQ и размеры мозга Зависит ли уровень интеллекта от размера головного мозга? (Willerman et al. 1991) .pull-left[  <small>[Scan_03_11](https://flic.kr/p/c45eZ3) by bucaorg(Paul_Burnett) on Flickr</small> ] .pull-right[ Было исследовано 20 девушек и 20 молодых людей У каждого индивида измеряли: - вес - рост - размер головного мозга (количество пикселей на изображении ЯМР сканера) - Уровень интеллекта измеряли с помощью IQ тестов <small>Пример: Willerman, L., Schultz, R., Rutledge, J. N., and Bigler, E. (1991), "In Vivo Brain Size and Intelligence", Intelligence, 15, p.223--228. Данные: ["The Data and Story Library"](http://lib.stat.cmu.edu/DASL) Фото: [Scan\_03\_11](https://flic.kr/p/c45eZ3) by bucaorg (Paul Burnett) on Flickr </small> ] --- ## Вспомним, на чем мы остановились ``` Call: lm(formula = PIQ ~ MRINACount, data = brain) Residuals: Min 1Q Median 3Q Max -39.6 -17.9 -1.6 17.0 42.3 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) 1.7437570 42.3923825 0.04 0.967 MRINACount 0.0001203 0.0000465 2.59 0.014 * --- Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 Residual standard error: 21 on 38 degrees of freedom Multiple R-squared: 0.15, Adjusted R-squared: 0.127 F-statistic: 6.69 on 1 and 38 DF, p-value: 0.0137 ``` --- ## Уравнение и график зависимости `$$PIQ_i = 1.744 + 0.0001202 \cdot MRINACount_i$$` <!-- --> --- class: middle, center, inverse # Тестирование гипотез о линейных моделях --- ## Способы проверки значимости модели и ее коэффициентов Два равноправных способа -- ### Значима ли модель целиком? + F критерий <!-- : действительно ли объясненная моделью дисперсия `\(MS_{r}\)` больше, чем случайная (= остаточная) дисперсия `\(MS_{e}\)`? --> -- ### Значима ли связь между предиктором и откликом? + t-критерий <!-- : отличается ли от нуля коэффициент `\(b_k\)` при этом предикторе `\(x_k\)` --> + F-критерий <!-- : действительно ли объясненная предиктором дисперсия `\(MS_{x_{k}}\)` больше, чем случайная (= остаточная) дисперсия `\(MS_{e}\)`? --> --- class: middle, center, inverse # Тестирование гипотез <br/>с помощью t-критерия --- ## Тестирование гипотез с помощью t-критерия ### Гипотезы Зависимость есть, если `\(\beta_k \ne 0\)` Нулевая гипотеза `\(H_0: \beta_k = 0\)` --- ## Тестирование гипотез с помощью t-критерия ### Гипотезы Зависимость есть, если `\(\beta_k \ne 0\)` Нулевая гипотеза `\(H_0: \beta_k = 0\)` ### Тестовая статистика `$$t=\frac{b_k - \beta_k}{SE_{b_k}} = \frac{b_k - 0}{SE_{b_k}} = \frac{b_k}{SE_{b_k}}$$` Число степеней свободы: `\(df = n − p\)`, где `\(n\)` — объем выборки, `\(p\)` — число параметров модели, `\(k\)` — конкретный коэффициент регрессии. Для простой линейной регрессии с одним предиктором `\(df = n - 2\)`. --- ## Зависит ли IQ от размера головного мозга? `$$PIQ_i = 1.744 + 0.0001202 \cdot MRINACount_i$$` ``` r summary(brain_model) ``` ``` Call: lm(formula = PIQ ~ MRINACount, data = brain) Residuals: Min 1Q Median 3Q Max -39.6 -17.9 -1.6 17.0 42.3 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) 1.7437570 42.3923825 0.04 0.967 MRINACount 0.0001203 0.0000465 2.59 0.014 * --- Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 Residual standard error: 21 on 38 degrees of freedom Multiple R-squared: 0.15, Adjusted R-squared: 0.127 F-statistic: 6.69 on 1 and 38 DF, p-value: 0.0137 ``` Результаты теста на IQ статистически значимо связаны <br/> с размерами мозга на МРТ ( `\(t_{ 0.05, 38}\)` = 2.59, `\(p\)` = 0.01 ). --- class: middle, center, inverse # Тестирование гипотез <br/>при помощи F-критерия --- ## Общая изменчивость Общая изменчивость `\(SS_{t}\)` — это сумма квадратов отклонений наблюдаемых значений `\(y_i\)` от общего среднего `\(\bar y\)` <!-- --> --- ## Структура общей изменчивости `$$SS_t = \color{blue}{SS_r} + \color{green}{SS_e}$$` <!-- --> --- ## От изменчивостей к дисперсиям <!-- --> | `\(MS_t\)`, полная дисперсия | `\(\color{blue}{MS_r}\)`, дисперсия, <br /> объясненная регрессией | `\(\color{green}{MS_e}\)`, остаточная дисперсия | |-----|-----|-----| | `\(MS_{t} =\frac{SS_{t}}{df_{t}}\)` | `\(\color{blue}{MS_{r}} =\frac{\color{blue}{SS_{r}}}{df_{r}}\)` | `\(\color{green}{MS_{e}} =\frac{\color{green}{SS_{e}}}{df_{e}}\)` | | `\(SS_{t}=\sum{(y_i - \bar{y})^2}\)` | `\(\color{blue}{SS_{r}}=\sum{(\hat{y}-\bar{y})^2}\)` | `\(\color{green}{SS_{e}}=\sum{(y_i - \hat{y})^2}\)` | | `\(df_{t} = n-1\)` | `\(df_{r} = 1\)` | `\(df_{e} = n-2\)` | --- ## С помощью `\(MS_r\)` и `\(MS_e\)` можно тестировать значимость коэффициентов Если дисперсии остатков для всех значений x равны, то `\(E(\color{blue}{MS_r}) = \color{green}{\sigma^2} + \color{red}{\beta_1}^2\sum(x_i - \bar x)^2\)` -- `\(= \color{green}{\sigma^2} + \sigma_{x}^2\)` `\(E(\color{green}{MS_e}) = \color{green}{\sigma^2}\)` <br/> -- Если зависимости нет, то `\(\color{red}{\beta_1} = 0\)`, и тогда -- `\(\color{blue}{MS_r} \approx \color{green}{MS_e}\)` <br/> -- Мы хотим протестировать гипотезы: - `\(H_0: \color{red}{\beta_1} = 0\)` - `\(H_A: \color{red}{\beta_1} \ne 0\)` Если справедлива `\(H_A: \color{red}{\beta_1} \ne 0\)`, то `\(\color{blue}{MS_r} > \color{green}{MS_e}\)` -- `$$F= \frac{\color{blue}{MS _{r}}}{\color{green}{MS_{e}}}$$` -- Поздравляю, вы только что изобрели _F_-критерий! --- ## Тестирование значимости коэффициентов регрессии при помощи F-критерия .pull-left-60[ - `\(H_0: \color{red}{\beta_1} = 0\)` - `\(H_A: \color{red}{\beta_1} \ne 0\)` `$$F_{df_r, df_e}= \frac{\color{blue}{MS _{r}}}{\color{green}{MS_{e}}}$$` Для простой линейной регрессии `\(df_{r} = 1\)` и `\(df_{e} = n - 2\)` <!-- --> ] .pull-right-40[ - У F-распределения два параметра: `\(df_{r}\)` и `\(df_{e}\)` - Логика F-теста аналогична t-критерию: Определяем вероятность того, что при *условии справедливости `\(H_0\)`* при повторных выборках появится значение F больше или равное F, полученному в нашем эксперименте. - F-тест будет односторонним, т.к. соотношение дисперсий может быть только положительным. ] --- ## Таблица результатов дисперсионного анализа | Источник изменчивости | df | SS | MS | F | | ----- | ----- | ----- | ----- | ----- | | .blue[Регрессия] | `\(df _r = 1\)` | `\(\color{blue}{SS _r} = \sum{(\hat y _i - \bar y)^2}\)` | `\(\color{blue}{MS _r} = \frac{\color{blue}{SS _r}}{df _r}\)` | `\(F _{df _r, df _e} = \frac{\color{blue}{MS _r}}{\color{green}{MS _e}}\)` | | .green[Остаточная]| `\(df _e = n - 2\)` | `\(\color{green}{SS _e} = \sum{(y _i - \hat y _i)^2}\)` | `\(\color{green}{MS _e} = \frac{\color{green}{SS _e}}{df _e}\)` | | Общая | `\(df _t = n - 1\)` | `\(SS _t = \sum {(y _i - \bar y)^2}\)` | <br/><br/> Минимальное упоминание результатов в тексте должно содержать `\(F _{df _r, df _e}\)` и `\(p\)`. --- class: middle, center, inverse # Оценка качества подгонки модели --- ## В чем различие между этими двумя моделями? <!-- --> -- У этих моделей разный разброс остатков: - Модель слева объясняет практически всю изменчивость - Модель справа объясняет не очень много изменчивости --- ## Коэффициент детерминации — мера качества подгонки модели __Коэффициент детерминации__ описывает какую долю дисперсии зависимой переменной объясняет модель `$$R^2 = \frac{\color{blue}{SS_{r}}}{SS_{t}}$$` - `\(0 < R^2 < 1\)` - `\(R^2 = r^2\)` — для простой линейной регрессии коэффициент детерминации равен квадрату коэффициента Пирсоновской корреляции --- ## Если в модели много предикторов, нужно внести поправку __Скорректированный коэффициет детерминации__ (adjusted R-squared) Применяется если необходимо сравнить две модели с разным количеством параметров $$ R^2_{adj} = 1- (1-R^2)\frac{n-1}{n-p}$$ `\(p\)` - количество параметров в модели Вводится штраф за каждый новый параметр --- ## Еще раз смотрим на результаты регрессионного анализа зависимости IQ от размеров мозга ``` r summary(brain_model) ``` ``` Call: lm(formula = PIQ ~ MRINACount, data = brain) Residuals: Min 1Q Median 3Q Max -39.6 -17.9 -1.6 17.0 42.3 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) 1.7437570 42.3923825 0.04 0.967 MRINACount 0.0001203 0.0000465 2.59 0.014 * --- Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 Residual standard error: 21 on 38 degrees of freedom Multiple R-squared: 0.15, Adjusted R-squared: 0.127 F-statistic: 6.69 on 1 and 38 DF, p-value: 0.0137 ``` -- Модель зависимости IQ от размеров мозга на МРТ объясняет всего лишь 15 % общей изменчивости IQ в этих данных. Ценность такой модели сомнительна. --- ## Как записываются результаты регрессионного анализа в тексте статьи? Мы показали, что связь между результатами теста на IQ и размером головного мозга на МРТ описывается моделью вида <br/> IQ = 1.74 + 0.00012 MRINACount ( `\(F_{1,38}\)` = 6.686, p = 0.014, `\(R^2\)` = 0.15) -- Пишем статью? --- ## Задание 1 & 2 Выполните задания 1 и 2 в одном из этих файлов: - 07_task_assumptions_catsM.R - 07_task_assumptions_GAG.R --- class: middle, center, inverse # Зачем нужна диагностика <br/>линейных моделей --- ## Если стохастическая модель адекватна, то что мы можем сказать про остатки? <!-- --> -- - Остатки --- это просто шум. Обычно считается, что `\(\varepsilon_i \in N(0, \sigma)\)`. - В остатках не должно быть никаких закономерностей (паттернов). --- ## Зачем нужна диагностика модели? <br/>Разве тестов было недостаточно? Пусть у нас есть некоторая модель, которая показывает зависимость `\(V_1 = f(V_2, V_3, V_4, V_5)\)` ``` r dat <- read.table('data/orly_owl_Lin_4p_5_flat.txt') fit <- lm(V1 ~ V2 + V3 + V4 + V5, data = dat) summary(fit) ``` ``` Call: lm(formula = V1 ~ V2 + V3 + V4 + V5, data = dat) Residuals: Min 1Q Median 3Q Max -2.065 -0.988 0.142 0.796 1.956 Coefficients: Estimate Std. Error t value Pr(>|t|) (Intercept) 0.00119 0.02117 0.06 0.96 V2 0.98633 0.12863 7.67 2.6e-14 *** V3 0.97229 0.12751 7.63 3.5e-14 *** V4 0.86143 0.12043 7.15 1.1e-12 *** V5 0.92747 0.08443 10.98 < 2e-16 *** --- Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 Residual standard error: 1 on 2293 degrees of freedom Multiple R-squared: 0.05, Adjusted R-squared: 0.0483 F-statistic: 30.2 on 4 and 2293 DF, p-value: <2e-16 ``` Все значимо! Пишем статью? --- ## Остатки модели Постройте график зависимости остатков от предсказанных значений при помощи этого кода ``` r library(car) residualPlot(fit, pch = ".") ``` --- ## Oh, really? ``` r library(car) residualPlot(fit, pch = ".") ``` <!-- --> [http://www4.stat.ncsu.edu/~stefanski/NSF_Supported/Hidden_Images/stat_res_plots.html](http://www4.stat.ncsu.edu/~stefanski/NSF_Supported/Hidden_Images/stat_res_plots.html) -- В остатках скрыта информация! --- ## Анализ остатков линейных моделей Этот анализ дает возможность: 1) Проверить данные на наличие влиятельных наблюдений и выявить их; 2) Проверить соответствует ли построенная модель условиям применимости линейных моделей. --- ## Условия применимости линейных моделей 1. Линейная связь 1. Независимость наблюдений 1. Соответствие нормальному распределению 1. Гомогенность дисперсий 1. Отсутствие коллинеарности предикторов (для множественной регрессии) --- class: middle, center, inverse # Анализ остатков --- ## Какие бывают остатки? -- ### "Сырые" остатки `$$\color{green}{e_i} = y_i - \color{blue}{\hat{y_i}}$$` -- ### Стандартизованные (стьюдентизированные) остатки `$$s_i = \frac{\color{green}{e_i}}{\sqrt{MS_e(1-\color{purple}{h_{ii}})}}$$` - легко сравнивать (стандартизованы), учитывают силу влияния наблюдений <hr /> - `\(\sqrt{MS_e}\)` — стандартное отклонение остатков - `\(\color{purple}{h_{ii}}\)` — "сила воздействия" отдельных наблюдений (leverage, рычаг проекционной матрицы) --- ## Что такое проекционная матрица? По определению, остатки `\(\color{green}{\mathbf{e}} = \mathbf{Y} - \color{blue}{\hat{\mathbf{Y}}}\)`. Тогда `\(\color{green}{\mathbf{e}} = \mathbf{Y} - \mathbf{X} \mathbf{b} = \mathbf{Y} - \mathbf{X}[\mathbf{X}'\mathbf{X}]^{-1}\mathbf{X}'\mathbf{Y}\)`. <br/> Обозначим `\(\color{purple}{\mathbf{H}} \equiv \mathbf{X}[\mathbf{X}'\mathbf{X}]^{-1}\mathbf{X}'\)`. Матрица `\(\color{purple}{\mathbf{H}}\)` — называется __"хэт"-матрица__ (_hat-matrix_) или __проекционная матрица__, т.к. она позволяет получить предсказанные значения из наблюдаемых. `\(\color{blue}{\hat{\mathbf{Y}}} = \color{purple}{\mathbf{H}}\mathbf{Y}\)` <br/> Тогда остатки можно получить как `\(\color{green}{\mathbf{e}} = \mathbf{Y} - \color{purple}{\mathbf{H}}\mathbf{Y} = (\mathbf{I} - \color{purple}{\mathbf{H}})\mathbf{Y}\)`. Диагональные элементы проекционной матрицы `\(\color{purple}{h_{ii}}\)` — это мера __воздействия__ точек на ход линии регрессии. --- class: middle, center, inverse # Влиятельные наблюдения --- ## Влиятельные наблюдения (influential points) Влиятельные наблюдения — это наблюдения, которые вносят слишком большой вклад в оценку параметров (коэффициентов) модели.  <small>Из кн. Quinn, Keough, 2002</small> Учет каких из этих точек повлияет на ход регрессии и почему? -- - Точка 1 почти не повлияет, т.к. у нее маленький остаток, хоть и экстремальное значение `\(X\)` - Точка 2 почти не повлияет, т.к. ее `\(X\)` близок к среднему, хоть и большой остаток - Точка 3 повлияет сильно, т.к. у нее не только большой остаток, но и экстремальное значение `\(X\)` --- ## Воздействие точек (leverage) __Воздействие__ точек `\(\color{purple}{h_{ii}}\)` (_leverage_) показывает силу влияния значений `\(x_i\)` на ход линии регрессии, то есть на `\(\color{blue}{\hat{y_i}}\)` .pull-left[  <small>Из кн. Quinn, Keough, 2002</small> ] .pull-right[  <small>Weighing Machine by neys fadzil on Flickr</small> ] -- Точки, располагающиеся дальше от `\(\bar{x}\)`, оказывают более сильное влияние на `\(\color{blue}{\hat{y_i}}\)` - `\(\color{purple}{h_{ii}}\)` варьирует в промежутке от `\(1/n\)` до 1 - Если `\(\color{purple}{h_{ii}} > 2(p/n)\)`, то надо внимательно посмотреть на данное значение ( `\(p\)` — число параметров модели, `\(n\)` — объем выборки) --- ## Расстояние Кука (Cook's distance) описывает, как повлияет на модель удаление данного наблюдения `$$D_i = \frac{\sum{( \color{blue}{\hat{y_{j}}} - \color{red}{\hat{y}_{j(i)}})^2}}{p \; MS_e}$$` - `\(\color{blue}{\hat{y_j}}\)` — значение предсказанное полной моделью - `\(\color{red}{\hat{y}_{j(i)}}\)` — значение, предсказанное моделью, построенной без учета `\(i\)`-го значения предиктора - `\(p\)` — количество параметров в модели - `\(MS_{e}\)` — среднеквадратичная ошибка модели ( `\(\hat\sigma^2\)` ) --- ## Расстояние Кука покажет оба аспекта влиятельности Формулу расстояния Кука для данного `\(i\)`-того наблюдения можно расписать: `$$D_i = \frac{\color{green}{e_i}^2}{p \; MS_e} \cdot \frac { \color{purple}{h_{ii}} } {(1 - \color{purple}{h_{ii}}) ^ 2}$$` Видно, что `\(D_i\)` зависит сразу от - `\(\color{green}{e_i}\)` — это величина остатков - `\(\color{purple}{h_{ii}}\)` — это воздействие наблюдений При каком значении `\(D_i\)` будем считать наблюдение выбросом? Два варианта: - `\(D_i > 1\)` — "мягкий" порог (для простоты в этом курсе такой) - `\(D_i > \frac{4}{(n - p)}\)` — "жесткий" порог ( `\(n\)` — объем выборки, `\(p\)` — число параметров) --- ## Что делать с наблюдениями-выбросами? -- - Удалить? -- __Осторожно!__ Только очевидные ошибки в наблюдениях можно удалять. Лучше найти причины. -- - Трансформировать? Это не всегда поможет. - Иногда можно переформулировать модель. --- ## Некоторые виды трансформаций Трансформация | Формула ------------- | ------------- степень -2 | `\(1/x^2\)` степень -1 | `\(1/x\)` степень -0.5 | `\(1/\sqrt{x}\)` степень 0.5 | `\(\sqrt{x}\)` логарифмирование | `\(log(x)\)` --- ## Данные для анализа остатков ``` r library(ggplot2) brain_diag <- fortify(brain_model) head(brain_diag, 2) ``` ``` PIQ MRINACount .hat .sigma .cooksd .fitted .resid .stdresid 1 124 816932 0.06638 20.88 0.0498380 99.98 24.017 1.1840 2 124 1001121 0.06687 21.27 0.0003039 122.13 1.868 0.0921 ``` - `.hat` — "сила воздействия" данного наблюдения (leverage) - `.cooksd` — расстояние Кука - `.fitted` — предсказанные значения - `.resid` — остатки - `.stdresid` — стандартизованные остатки --- ## График расстояния Кука Проверяем наличие влиятельных наблюдений в `brain_model`. Порядок значений на графике такой же как в исходных данных. ``` r # График расстояния Кука ggplot(brain_diag, aes(x = 1:nrow(brain_diag), y = .cooksd)) + geom_bar(stat = "identity") ``` <!-- --> -- - Есть одно влиятельное наблюдение, которое нужно проверить, но сила его влияния невелика (расстояние Кука `\(< 1\)`, и только одно наблюдение больше `\(4/(n-p) = 0.11\)`) --- ## График остатков от предсказанных значений Большую часть того, что нужно знать про остатки вы увидите на этом графике. А сейчас давайте научимся читать такой график. ``` r gg_resid <- ggplot(data = brain_diag, aes(x = .fitted, y = .stdresid)) + geom_point() + geom_hline(yintercept = 0) gg_resid ``` <!-- --> --- class: middle, center, inverse # 1. Линейность связи --- ## Нелинейность связи видно на графиках остатков <!-- --> __Проверка на линейность связи__ - График зависимости `\(y\)` от `\(x\)` (и от других переменных, не включенных в модель) - График остатков от предсказанных значений --- ## Что делать, если связь нелинейна? - Добавить неучтенные переменные или взаимодействия; - Применить линеаризующее преобразование (Осторожно!); - Применить обобщенную линейную модель с другой функцией связи (GLM); - Построить аддитивную модель (GAM), если достаточно наблюдений по `\(x\)`; - Построить нелинейную модель, если известна форма зависимости. --- ## Примеры линеаризующих преобразований __Логарифмирование__ <!-- --> __Возведение в степень__, и т.д. <!-- --> --- ## Не стоит трансформировать отклик! <!-- --> __Осторожно!__ Вы рискуете изучить не то, что хотели: 1.При логарифмировании отклика вы будете изучать поведение мат.ожидания логарифма `\(E(log(y_i)) = b_0 + b_1x_{1i} + ... + e_i\)`. 2.Трансформация отклика не только линеаризует зависимость, но и затронет величину остатков `\(e\)`. -- Вместо трансформации отклика лучше использовать обобщенную линейную модель с подходящей функцией связи, например: `\(log(E(y_i)) = b_0 + b_1x_{1i}+ ... + e_i\)` --- class: middle, center, inverse # 2. Независимость --- ## Каждое значение `\(y_i\)` должно быть независимо от любого другого `\(y_j\)` Это нужно контролировать на этапе планирования сбора материала * Наиболее частые источники зависимостей: + псевдоповторности (повторно измеренные объекты) + неучтенные переменные + временные автокорреляции (если данные - временной ряд) + пространственные автокорреляции (если пробы взяты в разных местах) + и т.п. --- ## Диагностика нарушений независимости Взаимозависимости можно заметить на графиках остатков - остатки vs. предсказанные значения - остатки vs. переменные в модели - остатки vs. переменные не в модели --- ## Нарушение независимости: Неучтенная переменная 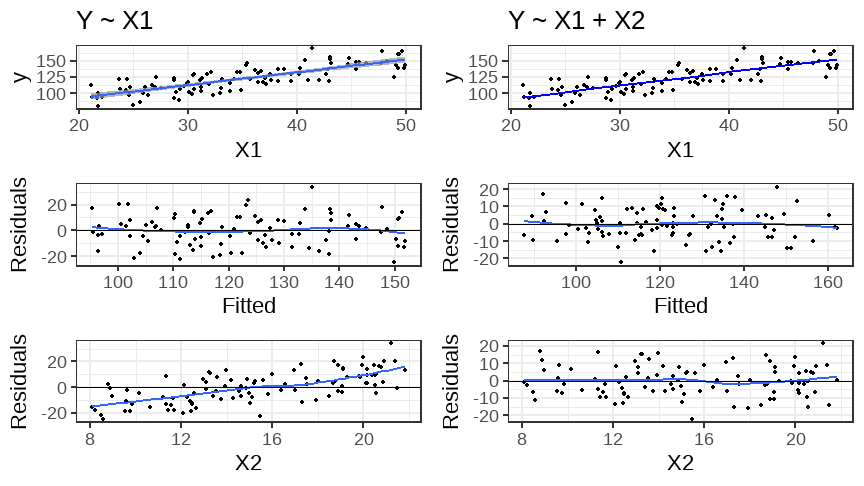<!-- --> -- - Слева: Если в модели не учтена переменная `\(X2\)`, внешне все нормально, но величина остатков зависит от `\(X2\)` - Справа: Если `\(X2\)` учесть, то зависимость остатков от `\(X2\)` исчезает --- ## Нарушение независимости: Автокорреляция В данном случае, наблюдения — это временной ряд <br/>или ряд наблюдений в пространстве. 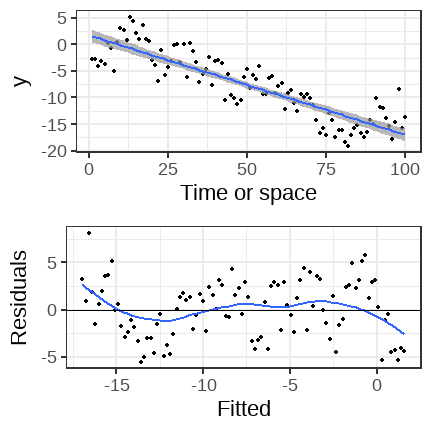<!-- --> -- На графиках остатков четко видно, что остатки не являются независимыми. Наблюдения, расположенные на определенном расстоянии друг от друга, имеют остатки похожей величины. --- ## Чем опасна автокорреляция (и любая другая зависимость наблюдений друг от друга) ``` r set.seed(123) population <- rnorm(1000) population_correlated <- arima.sim(list(order = c(1,0,0), ar = 0.5), n = 1000, sd=1) ``` 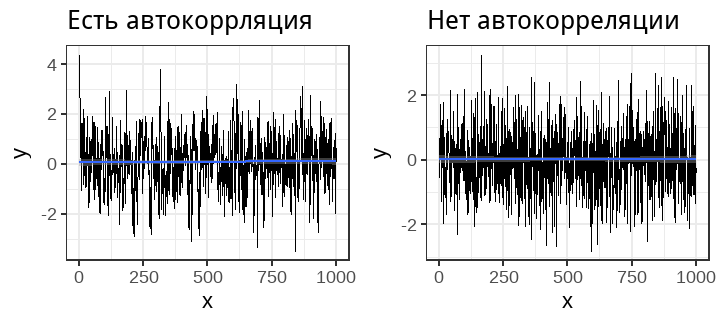<!-- --> --- ## Чем опасна автокорреляция (и любая другая зависимость наблюдений друг от друга) .pull-left-60[ Построим 1000 моделей на случайных выборках по 10 последовательных наблюдений ``` r p_values <- data.frame(p_no_cor = rep(NA, 1000), p_cor = NA) for(i in 1:1000){ begin = sample(1:900, 1) x = begin:(begin + 9) y_cor = population_correlated[begin:(begin + 9)] #Случайная выборка из генеральной совокупности с автокорреляцией y = population[begin:(begin + 9)] #Случайная выборка из генеральной совокупности без автокорреляции df = data.frame(x, y_cor, y) M_no_cor <- lm(y ~ x, data = df) M_cor <- lm(y_cor ~ x, data = df) p_values$p_no_cor[i] <- summary(M_no_cor)$coefficients[2, 4] #p-value из summary модели p_values$p_cor[i] <- summary(M_cor)$coefficients[2, 4] #p-value из summary модели } ``` ] -- .pull-right-40[ Для простого "шума", когда нет автокорреляций и нет связи между `\(y\)` и `\(x\)` (справедлива `\(H_0\)`) частота ошибок составляет ``` r mean(p_values$p_no_cor < 0.05) ``` ``` [1] 0.048 ``` ] -- .pull-right-40[ Для автокоррелированных данных, при отсутствии связи между `\(y\)` и `\(x\)` (справедлива `\(H_0\)`) частота ошибок составляет ``` r mean(p_values$p_cor < 0.05) ``` ``` [1] 0.233 ``` ] --- ## Проверка на автокорреляцию Проверка на автокорреляцию нужна, если данные --- это временной ряд, или если известны координаты или время сбора проб. Способы проверки временной автокорреляции (годятся, если наблюдения в ряду расположены через равные интервалы): - График автокорреляционной функции остатков (ACF-plot) покажет корреляции с разными лагами. 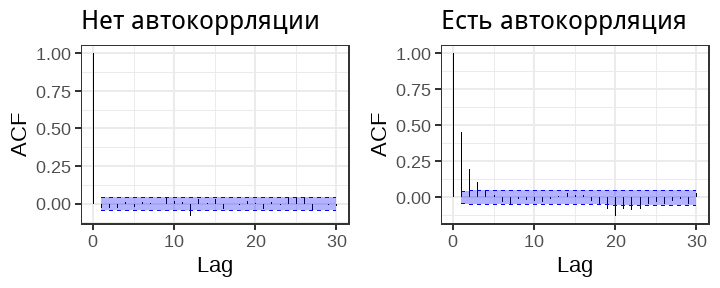<!-- --> - Критерий Дарбина-Уотсона (значимость автокорреляции 1-го порядка). Для проверки пространственных автокорреляций: *вариограммы* <!-- - I Морана (Moran's I) --> --- ## Что делать, если у вас нарушено условие независимости значений? Выбор зависит от обстоятельств. Вот несколько возможных вариантов. + псевдоповторности - избавляемся от псевдоповторностей, вычислив среднее - подбираем смешанную модель (модель со случайным фактором) + неучтенные переменные - включаем в модель (если возможно) + временные автокорреляции - моделируем автокорреляцию - подбираем модель со случайным фактором + пространственные автокорреляции - моделируем пространственную автокорреляцию - делим на пространственные блоки и подбираем модель со случайным фактором (= random effects model, mixed model) --- ## Проверка условия независимости ### Графики зависимости остатков от предикторов в модели ``` r # Полный код ggplot(data = brain_diag, aes(x = MRINACount, y = .stdresid)) + geom_point() + geom_hline(yintercept = 0) ``` <!-- --> ``` r # То же самое с использованием ранее созданного gg_resid gg_resid + aes(x = MRINACount) ``` --- ### Графики зависимости остатков от предикторов не в модели В данном случае их нет --- class: middle, center, inverse # 3. Нормальное распределение --- ## 3. Нормальное распределение `\(y\)` <br/>(для каждого уровня значений `\(x\)`) .pull-left-60[ <!-- --> ] .pull-right-40[ Это условие невозможно проверить "в лоб", т.к. обычно каждому `\(x\)` соответствует лишь небольшое число `\(y\)` ] --- ## Проверка нормальности распределения остатков Если `\(y\)` это нормально распределенная случайная величина `$$y_i \sim N(\mu_{y_i}, \sigma)$$` -- и мы моделируем ее как `$$y_i = b_0 + b_1x_{1i} + \cdots + e_i$$` -- то остатки от этой модели — тоже нормально распределенная случайная величина `$$e_i \sim N(0, \sigma)$$` -- Т.е. выполнение этого условия можно оценить по поведению случайной части модели. --- ## Проверка нормальности распределения остатков Есть формальные тесты, но: - у формальных тестов тоже есть свои условия применимости - при больших выборках формальные тесты покажут, что значимы даже небольшие отклонения от нормального распределения - тесты, которые используются в линейной регрессии, устойчивы к небольшим отклонениям от нормального распределения -- Лучший способ проверки — квантильный график остатков. --- ## Квантильный график остатков По оси Х — квантили теоретического распределения, по оси Y — квантили остатков модели. Если наблюдаемое распределение соответствует теоретическому, то точки должны лечь вдоль прямой по диагонали графика. <br/> -- Обычные остатки должны подчиняться нормальному распределению `\(e \sim N(0, \sigma)\)` -- Cтьюдентизированные остатки — t-распределению ``` r # library(car) qqPlot(brain_model, id = FALSE) # из пакета car ``` <!-- --> --- ## Что делать, если остатки распределены не нормально? Зависит от причины - Нелинейная связь? - Построить аддитивную модель (если достаточно наблюдений по `\(x\)`) - Построить нелинейную модель (если известна форма зависимости) - Неучтенные переменные? - добавляем в модель - Зависимая переменная распределена по-другому? - трансформируем данные (неудобно) - подбираем модель с другим распределением остатков (обобщенную линейную модель) --- class: middle, center, inverse # 4. Постоянство дисперсии --- ## 4. Постоянство дисперсии <br/>(= гомогенность дисперсии, гомоскедастичность) .pull-left-60[ <!-- --> ] .pull-right-40[ Это самое важное условие, поскольку многие тесты чувствительны к гетероскедастичности. ] --- ## Проверка гомогенности дисперсий Есть формальные тесты (тест Бройша-Пагана, тест Кокрана), но: - у формальных тестов тоже есть свои условия применимости, и многие сами неустойчивы к гетероскедастичности - при больших выборках формальные тесты покажут, что значима даже небольшая гетероскедастичность -- Лучший способ проверки на гомогенность дисперсий — график остатков от предсказанных значений. --- ## Проверка гомогенности дисперсий <!-- --> --- ## Чем опасна гетероскедастичность Создадим две генеральные совокупности, в которых нет связи между `\(y\)` и `\(x\)` <!-- --> --- ## Чем опасна гетероскедастичность .pull-left-60[ Возьмем 1000 выборок из этих совокупностей по 10 наблюдений и построим много линейных моделей ``` r p_values <- data.frame(p_no_heter = rep(NA, 1000), p_heter = NA) for(i in 1:1000){ df <- dat2[sample(1:1000, 10), ] M_no_heter <- lm(y_1 ~ x, data = df) M_heter <- lm(y_2 ~ x, data = df) p_values$p_no_heter[i] <- summary(M_no_heter)$coefficients[2, 4] #p-value из summary модели p_values$p_heter[i] <- summary(M_heter)$coefficients[2, 4] #p-value из summary модели } ``` ] -- .pull-right-40[ - Нет гетероскедастичности и нет связи между `\(y\)` и `\(x\)` (справедлива `\(H_0\)`) частота ошибок составляет ``` r mean(p_values$p_no_heter < 0.05) ``` ``` [1] 0.039 ``` ] .pull-right-40[ - Для совокупности с гетероскедастичностью, при отсутствии связи между `\(y\)` и `\(x\)` (справедлива `\(H_0\)`) частота ошибок составляет ``` r mean(p_values$p_heter < 0.05) ``` ``` [1] 0.073 ``` ] --- ## Проверка на гетероскедастичность Этот график у нас уже есть ``` r gg_resid ``` <!-- --> -- - Гетерогенность дисперсий не выражена. --- ## Что делать, если найдена гетероскедастичность? <!-- --> -- Трансформация может помочь, но лучше изменить модель. --- ## Возможные причины гетероскедастичности Даже если трансформация может помочь, лучше поискать причину гетерогенности дисперсий - Неучтенные переменные - добавляем в модель - Зависимая переменная распределена по-другому - трансформируем данные (неудобно) - подбираем модель с другим распределением остатков (обобщенную линейную модель) - Моделируем гетерогенность дисперсии. --- class: middle, center, inverse # Тренинг по анализу остатков --- ## Некоторые частые паттерны на графиках остатков .pull-left-60[  .tiny[Из кн. Logan, 2010, стр. 174] ] -- .pull-right-40[ - Рис. a — Условия применимости соблюдаются, модель хорошая - Рис. b — Клиновидный паттерн. Есть гетероскедастичность. Модель плохая - Рис. c — Остатки рассеяны равномерно, но нужны дополнительные предикторы - Рис. d — Нелинейный паттерн. Линейная модель использована некорректно ] --- ## Задание 3 Выполните три блока кода Какие нарушения условий применимости линейных моделей здесь наблюдаются? Вам понадобятся 1. График расстояния Кука 2. График остатков от предсказанных значений 3. Графики остатков от предикторов в модели и не в модели 4. Квантильный график остатков --- ## Задание 3, блок 1 ``` r set.seed(90829) x1 <- seq(1, 100, 1) y1 <- diffinv(rnorm(99)) + rnorm(100, 0.2, 4) dat1 = data.frame(x1, y1) ggplot(dat1, aes(x = x1, y = y1)) + geom_point()+ geom_smooth(method="lm", alpha = 0.7) ``` <!-- --> --- ## Решение, блок 1 ### Графики <!-- --> -- - Выбросов нет, зависимость нелинейна - Небольшие отклонения от нормального распределения --- ## Решение, блок 1 ``` r mod1 <- lm(y1 ~ x1, data = dat1) # Данные для графиков остатков mod1_diag <- fortify(mod1) # 1) График расстояния Кука ggplot(mod1_diag, aes(x = 1:nrow(mod1_diag), y = .cooksd)) + geom_bar(stat = "identity") # 2) График остатков от предсказанных значений gg_resid <- ggplot(data = mod1_diag, aes(x = .fitted, y = .stdresid)) + geom_point() + geom_hline(yintercept = 0) gg_resid # 3) Графики остатков от предикторов в модели и не в модели gg_resid + aes(x = x1) # 4) Квантильный график остатков qqPlot(mod1, id = FALSE) ``` --- ## Задание 3, блок 2 ``` r set.seed(7657674) x2 <- runif(1000, 1, 100) b_0 <- 100; b_1 <- -20 h <- function(x) x^0.5 eps <- rnorm(1000, 0, h(x2)) y2 <- b_0 + b_1 * x2 + eps dat2 <- data.frame(x2, y2) ggplot(dat2, aes(x = x2, y = y2)) + geom_point() + geom_smooth(method = "lm") ``` <!-- --> --- ## Решение, блок 2 <!-- --> -- - Выбросов нет - Гетерогенность дисперсий, остатки не подчиняются нормальному распределению --- ## Решение, блок 2 ``` r mod2 <- lm(y2 ~ x2, data = dat2) # Данные для графиков остатков mod2_diag <- fortify(mod2) # 1) График расстояния Кука ggplot(mod2_diag, aes(x = 1:nrow(mod2_diag), y = .cooksd)) + geom_bar(stat = "identity") # 2) График остатков от предсказанных значений gg_resid <- ggplot(data = mod2_diag, aes(x = .fitted, y = .stdresid)) + geom_point() + geom_hline(yintercept = 0) gg_resid # 3) Графики остатков от предикторов в модели и не в модели gg_resid + aes(x = x2) # 4) Квантильный график остатков qqPlot(mod2, id = FALSE) ``` --- ## Задание 3, блок 3 ``` r set.seed(9283) x3 <- rnorm(25, 50, 10) b_0 <- 20; b_1 <- 20; eps <- rnorm(50, 0, 100) y3 <- b_0 + b_1*x3 + eps y3[100] <- 1000; x3[100] <- 95; y3[99] <- 1300; x3[99] <- 90; y3[98] <- 1500; x3[98] <- 80 dat3 <- data.frame(x3, y3) ggplot(dat3, aes(x=x3, y=y3)) + geom_point() + geom_smooth(method="lm") ``` <!-- --> --- ## Решение, блок 3 <!-- --> -- - 100-е наблюдение сильно влияет на ход регрессии - Зависимость может быть нелинейна --- ## Решение, блок 3 ``` r mod3 <- lm(y3 ~ x3, data = dat3) # Данные для графиков остатков mod3_diag <- fortify(mod3) # 1) График расстояния Кука ggplot(mod3_diag, aes(x = 1:nrow(mod3_diag), y = .cooksd)) + geom_bar(stat = "identity") # 2) График остатков от предсказанных значений gg_resid <- ggplot(data = mod3_diag, aes(x = .fitted, y = .stdresid)) + geom_point() + geom_hline(yintercept = 0) gg_resid # 3) Графики остатков от предикторов в модели и не в модели gg_resid + aes(x = x3) # 4) Квантильный график остатков qqPlot(mod3, id = FALSE) ``` --- ## Задание 4 Выполните последнее задание в одном из этих файлов: - 07_task_assumptions_catsM.R - 07_task_assumptions_GAG.R --- ## Take-home messages - Гипотезы о наличии зависимости можно тестировать при помощи t- или F-теста. - Качество подгонки модели можно оценить при помощи коэффициента детерминации `\(R^2\)`. -- - У линейных моделей есть условия применимости, поэтому не спешите описывать результаты — сначала проверьте. -- - Если условия применимости нарушены, то результатам тестов для этой модели нельзя верить (получаются заниженные доверительные вероятности, возрастает вероятность ошибок I рода). - Анализ остатков дает разностороннюю информацию о валидности моделей. --- ## Что почитать + Гланц, С., 1998. Медико-биологическая статистика. М., Практика + Кабаков Р.И. R в действии. Анализ и визуализация данных на языке R. М.: ДМК Пресс, 2014 + Diez, D.M., Barr, C.D. and Çetinkaya-Rundel, M., 2015. OpenIntro Statistics. OpenIntro. + Zuur, A., Ieno, E.N. and Smith, G.M., 2007. Analyzing ecological data. Springer Science & Business Media. + Quinn G.P., Keough M.J. 2002. Experimental design and data analysis for biologists + Logan M. 2010. Biostatistical Design and Analysis Using R. A Practical Guide